Intégration génomique
Des ARN chimériques homme-virus ont été observées dans des cellules humaines infectées par le SARS-CoV-2 in vitro et in vivo
Serait-il possible que le génome du virus SARS-CoV-2 s'intégre dans le génome humain ?
Résumé tout public
Des travaux publiés d'abord en décembre 2020 puis en avril 2021 par une équipe américaine ont jeté un trouble et alimenté une controverse dans la communauté scientifique internationale. Les résultats de ces travaux suggèrent que des parties du génome du SARS-CoV-2 (le virus responsable de la COVID-19) pourraient s'intégrer sous forme d'ADN dans le génome humain.
Le SARS-CoV-2 est un virus dont le génome est constitué d'ARN. Comment alors pourrait-on le retrouver dans le génome humain qui lui est constitué d'ADN ?
Le génome humain contient une ensemble de gènes appelés LINE-1 dont la fonction est de se propager dans le génome en "sautant" d'un chromosome à un autre sur un mode "copier-coller". De fait, ces gènes LINE-1 ont été appelés "gènes sauteurs" et contribuent pour près de 17% au génome humain. Ces gènes, nommés rétrotransposons, sont issus de vestiges d'infections très anciennes par des rétrovirus dont ils ont gardé la capacité à produire de l'ADN à partir de l'ARN, d'où le préfixe "rétro".
Pour l'équipe américaine qui a soulevé cette question, ce sont les rétrotransposons LINE-1 qui prennent en charge des ARN du virus SARS-CoV-2, les rétro transcrivent en ADN, et les intègrent au génome des cellules du malade. On retrouve alors dans les cellules ou les tissus infectés des ARN chimériques homme-virus. La présence de ces ARN chimériques pourrait expliquer pourquoi des personnes, guéries de leur infection et ayant respecté des périodes de quarantaine strictes, sont testées positives au génome viral par test RT-PCR.
La possibilité d'une intégration du génome viral au génome humain ne serait pas sans conséquences. Celles-ci pourraient être à la fois néfastes (perturbations de l'expression de gènes pouvant conduire à diverses pathologies dont des cancers) et bénéfiques (possibilité de conférer au malade une immunité spécifique au virus sur le long-terme).
Cependant, ce mécanisme est contesté par d'autres équipes de chercheurs sur la base de problèmes liés aux modèles cellulaires utilisés, aux approches expérimentales, à l'analyse et aux interprétations des résultats.
Il n'y a pour l'heure pas encore de consensus scientifique clair sur la proposition selon laquelle le génome du SARS-CoV-2 pourrait s'intégrer dans le génome humain.
Cependant, les antivaccins se sont rapidement emparés de ces résultats afin de propager l'idée FAUSSE selon laquelle les vaccins à ARN pourraient conduire à une intégration génomique de l'ARN codant pour la protéine S du SARS-CoV-2.
Dans les cellules infectées par le SARS-CoV-2, les ARN viraux sont continuellement synthétisés du fait de la multiplication active des virions et restent présents pour de longues périodes, entre plusieurs jours jusqu'à plusieurs mois. Ces ARN auraient donc largement le temps d'être rétro transcrits par les rétrotransposons LINE-1. Or, cela serait malgré tout extrêmement rare et reste pour l'heure hypothétique.
En revanche, dans les deux vaccins à ARN Pfizer et Moderna couramment utilisés aujourd'hui, l'ARN codant pour la protéine S du SARS-CoV-2 est administré en intramusculaire en très faible quantité (deux doses de 30 ou 100 microgrammes, respectivement). Le nombre des cellules "touchées" par le vaccin est très faible et leur temps d'exposition à l'ARN du vaccin très court comparé à la durée d'une infection (jours-semaines-mois). De plus, l'ARN du vaccin n'est pas reproduit dans les cellules mais au contraire rapidement dégradé dès que sa traduction en protéine S est terminée.
La probabilité que l'ARN vaccinal codant pour la protéine S soit rétro transcrit et intégré au génome des cellules infectées est donc extrêmement faible, pour ne pas dire nulle, ce que l'objectivité scientifique m'interdit de dire.
En tout état de cause, le danger potentiel que représente l'intégration d'un génome étranger dans le génome humain serait plus important chez le sujet infecté que chez le sujet vacciné. Enfin, il a été démontré que ce phénomène d'intégration génomique est possible pour d'autres virus.
Pour celles et ceux qui désirent aller plus loin sur cette question, lire la suite ci-dessous.
La possibilité de l'intégration du SARS-CoV-2 dans le génome humain est une question intéressante et importante qui, pour l'heure, est très débattue par les scientifiques. En effet l'intégration du génome viral du SARS-CoV-2 dans le génome humain pourrait avoir des conséquences biologiques et pathologiques importantes pour les personnes infectées. Nous allons examiner ici des résultats expérimentaux qui suggèrent cette possibilité ainsi que ceux qui ne la soutiennent pas.
Petits rappels
Le dogme central de la biologie moléculaire est le suivant:
ADN à ARN pré-m à ARNm à protéine
L'ADN (constituant des gènes contenus dans les chromosomes) est transcrit en ARN pré-messager (réplique exacte du gène); l'ARN pré-m subit une opération d'épissage au cours de laquelle les séquences introniques du gène sont éliminées et seules les séquences exoniques sont préservées; cette opération, comme la précédente, se passe dans le noyau des cellules et conduit à la synthèse de l'ARN messager (ARNm) qui sort du noyau et est traduit en protéine dans le cytoplasme de la cellule.
Les virus détournent la machinerie de la cellule hôte pour répliquer leurs génomes (ARN ou ADN) et créer leurs progénitures de virions qui vont à leur tour se répliquer afin d'être exportés vers d'autres cellules qu'ils vont pouvoir infecter. La forme extrême de parasitisme viral est l'intégration d'une copie du génome viral dans l'ADN de la cellule hôte. Diverses classes de virus à ARN sont capables de cette perversion en créant une copie d'ADN complémentaire (ADNc) par transcription inverse de leur génome au cours de leur cycle de vie. Cette copie ADN de leur génome est ensuite intégrée au hasard dans le génome de l'hôte. Ces virus appelés rétrovirus (par exemple le virus du SIDA) violent le dogme de la biologie moléculaire en fabriquant de l'ADN à partir de l'ARN. D'où le préfixe "rétro".
Rétrovirus: ARN à ADNc à intégration dans le génome (ADN) de la cellule hôte
L'opération ARN à ADNc est catalysée par des enzymes appelées transcriptases inverses ou rétrotranscriptases ou RT. L'ADN formé est la réplique exacte de l'ARN et est appelé ADN complémentaire (ADNc). Il ne contient pas de séquences introniques. La première rétrotranscriptase a été découverte dans le virus du sarcome de Rous.
L'intégration de l'ADNc viral dans l'ADN de l'hôte est une étape obligatoire caractéristique des rétrovirus, ainsi que des rétro éléments endogènes comme les rétrovirus endogènes (voir ICI sur ce site) et les rétrotransposons.
Le SARS-CoV-2 responsable de la présente pandémie de COVID-19 n'est pas un rétrovirus. Il n'a donc aucun moyen d'intégrer son génome à celui des personnes infectées.
Observations cliniques sur des cohortes de malades
Des tests RT-PCR positifs continus ou récurrents pour le SARS-CoV-2 ont été signalés chez des patients des semaines ou des mois après guérison de l'infection initiale. Bien que des cas de réinfection authentique par le SARS-CoV-2 après guérison aient été signalés récemment, des études de cohorte, avec mise en quarantaine stricte de sujets guéris de la COVID-19, suggèrent que les cas de "re-positivité" n'étaient pas dus à une réinfection authentique par le virus. De fait, aucun virus capable de se répliquer n'a été isolé ou s'est propagé à partir de ces patients "re-positifs".
Quelle serait donc la cause d'une telle production prolongée et récurrente d'ARN viral sans infection virale sous-jacente ?
Hypothèse
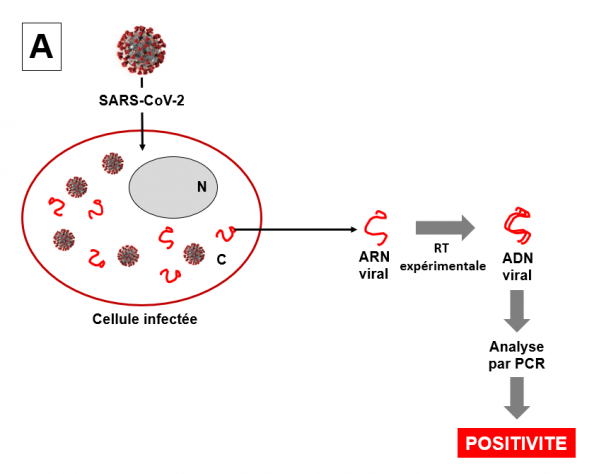
Dans l'immense majorité des cas, le virus entre dans les cellules et réplique son génome (ARN) qui est traduit en protéines par la machinerie cellulaire. De nouveaux virus sont produits et à leur tour se répliquent. La cellule contient ainsi de nombreuses copies des ARN viraux que l'on détecte par une analyse des cellules en RT-PCR (positivité du test PCR; voir PCR sur ce site). Voir figure ci-dessous.
Schéma très simplifié d'une analyse en RT-PCR
La cellule infectée contient des virions qui répliquent leur génome afin de produire de nouveaux virions. La cellule contient donc un grand nombre d'ARN viraux sous-génomiques, c'est à dire des ARN messagers viraux différentes codant pour des protéines virales différentes. L'ARN viral est représenté ici par un trait rouge. Par souci de simplification, un seul ARN viral est représenté. Pour réaliser une analyse en RT-PCT, la première étape consiste à récupérer des cellules infectées (prélèvements cliniques) et à en extraire les ARN totaux dont fait partie l'ARN viral. Dans un souci de simplification, l'ARN cellulaire largement majoritaire n'est pas représenté ici. La deuxième étape de l'analyse consiste à synthétiser expérimentalement les ADNc (ADN complémentaires, répliques des ARN) par utilisation d'une transcriptase inverse (RT) généralement issue de rétrovirus tels que le HIV. L'ADN est représenté par un double trait rouge, figurant les deux brins de l'ADN. Dans une troisième étape, les ADNc synthétisés sont amplifiés par PCR et quantifiés. Si la personne testée n'est pas infectée ou guérie, le résultat de la PCR est négatif, les cellules ne contenant pas de virus. C désigne le cytoplasme de la cellule, N le noyau.
Cependant, dans certains cas très rares, après guérison et quarantaine stricte, des tests en RT-PCR s'avèrent positifs pour le SARS-CoV-2. Cette situation pourrait résulter du fait que des fragments du génome viral (ARN) ont été rétro-transcrits en ADNc puis insérés dans le génome de l'hôte. La transcription (c'est à dire la production d'ARN) de copies d'ADN SARS-CoV-2 intégrées dans le génome des cellules précédemment infectées pourrait donc être responsable de tests PCR positifs longtemps après que l'infection initiale ait été éliminée. Voir figure ci-dessous.
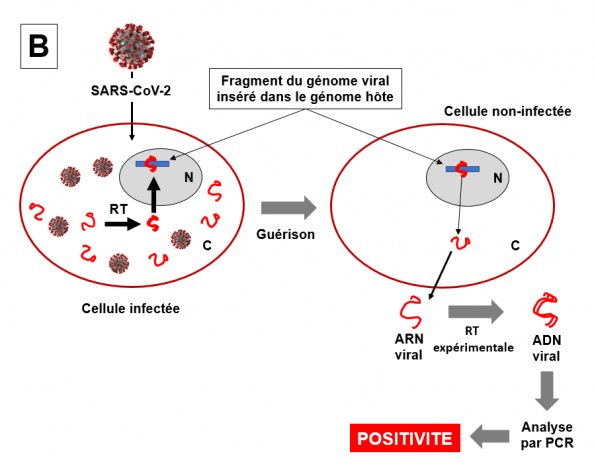
Hypothèse
A gauche une cellule infectée contenant des virions et des ARN viraux sous-génomiques en grand nombre (trait rouge). Les cellules humaines peuvent contenir des transcriptases inverses (RT) issues de télomères (extrémités des chromosomes) ou de rétrotransposons LINE-1 (voir le texte). Certains de ces ARN peuvent – très rarement - être pris en charge par les rétrotransposons LINE-1, être transformés en ADN et être intégrés dans un chromosome (rectangle bleu) dans le noyau cellulaire (N, ellipse en gris) par une ou plusieurs transcriptases inverses cellulaires (trait rouge en double, figurant les deux brins de l'ADN). Dans un souci de simplification, un seul chromosome est montré ici. Après plusieurs semaines ou plusieurs mois, la personne préalablement infectée a guéri. Ses cellules ne contiennent ni ne produisent de virions (cellule de droite). Cependant, le fragment d'ADN issu de l'ARN viral est toujours présent dans le chromosome. Cet ADN peut être transcrit en ARN. Si cette cellule est soumise à une analyse en RT-PCR elle sera positive malgré l'absence de virus. C désigne le cytoplasme de la cellule.
Explications
Virus à ARN à brin positif, le SARS-CoV-2 et d'autres bêta-coronavirus comme le SARS-CoV-1 et le MERS utilisent une ARN polymérase ARN-dépendante pour répliquer leur ARN génomique et transcrire leurs ARN sous-génomiques (voir sur ce site ICI). Le cycle de vie du SARS-CoV-2 ne comporte pas de phase nucléaire. Il est donc biologiquement peu probable qu'il se retrouve dans un endroit de la cellule où des événements d'épissage pourraient entraîner une intégration au génome.
Pour que l'ARN subisse une transcription inverse (passage de l'ARN à l'ADN) il faut qu'il soit pris en charge par des enzymes (transcriptases inverses) capables de réaliser cette opération. De telles enzymes sont absentes chez les coronavirus. Par contre, elles sont présentes chez les rétrovirus de type HIV, et chez l'homme au niveau des télomères (télomérase impliquée dans le maintien des extrémités des chromosomes) et dans divers rétrotransposons autonomes dont font partie les rétrovirus endogènes (voir sur ce site ici) et les éléments LINE-1 (dits "gènes sauteurs" ou "Long Interspersed Nuclear Element" en anglais). De fait, une activité de transcription inverse, bien que très faible, est observée dans les cellules humaines.
Il a déjà été montré que des ARN cellulaires subissent une transcription inverse et leur ADNc s'intègre dans le génome. C'est le cas par exemple du gène APP (amyloid-beta precursor protein) dont on retrouve des ADNc intégrés dans le génome des neurones; ces ADNc sont ensuite transcrits en ARN. Un autre exemple concerne des séquences de virus à ARN non rétroviraux comme le virus de la stomatite vésiculaire, le virus de la chorioméningite lymphocytaire, Bornavirus, Ebola virus et Marburg virus). Ces séquences ont été détectées dans les génomes de nombreuses espèces de vertébrés et plusieurs intégrations présentent des signaux compatibles avec l'implication des rétrotransposons LINE-1.
Il a donc été proposé que la détection par RT-PCR de séquences du SARS-CoV-2 dans des prélèvements de personnes guéries de leur infection résultent : i) d'une transcription inverse d'ARN sous-génomiques viraux ainsi transformés en fragments d'ADN viral par les rétrotransposons LINE-1, ii) intégration de ces fragments d'ADN viral dans le génome de l'hôte par les rétrotransposons LINE-1, et iii) transcription de ces fragments d'ADN viral en ARN détectés par les analyses RT-PCR. Du fait de l'intégration des fragments d'ADN viral dans le génome de l'hôte, on doit s'attendre à détecter des ARN chimériques homme-virus.
Dès sa proposition, cette possibilité a été fortement contestée par la communauté scientifique internationale et a fait l'objet de débats non encore clos. Nous allons examiner dans ce qui suit les arguments pour et les arguments contre cette proposition.
Les rétrotransposons humains LINE-1
Les rétrotransposons humains LINE-1 constituent près de 17% du génome. Ce sont des vestiges de très anciennes infections rétrovirales survenues au cours de l'évolution des espèces en général et des hominidés en particulier. Ce type de rétrotransposons est une source potentielle de transcriptase inverse endogène, capable de transposition inverse et intégration génomique de leurs propres gènes (en mode "copier-coller"; gène sauteurs) mais aussi d'autres éléments non autonomes tels que les séquences Alu et les ARN cellulaires ou viraux. Et c'est à cette dernière possibilité (rétro transposition d'ARN viraux) que nous allons nous intéresser ici. A noter que les éléments LINE-1 manifestent une forte préférence (d'un facteur compris entre 2000 et 10000) pour la rétro transposition de leur propre ARN par rapport à la rétro transposition d'autres ARN. Autrement dit, sur 10000 évènements de rétro transposition médiée par les éléments LINE-1, seulement entre 1 et 5 concernent d'autres ARN.
Les rétrotransposons LINE-1 pleine longueur sont capables de transposition inverse autonome. Ils se propagent en étant transcrits par l’ARN polymérase II à partir d’un promoteur interne, puis utilisent respectivement leur endonucléase et leur transcriptase inverse pour couper l'ADN sur un site cible (une séquence spécifique riche en T; 5'-TTTT/AA) et assurer la transcription inverse de leur ARN, en intégrant l'ADN codant pour l'élément LINE-1 dans un nouveau locus génomique. Ce processus est connu sous le nom de "transcription inverse amorcée par le site cible" (TPRT en anglais). Une nouvelle insertion de l'élément LINE-1 est généralement flanquée de courtes répétitions directes dérivées du locus d’ADN cible lors de l’intégration; ces répétitions sont appelées duplications de sites cibles (TSD en anglais) et peuvent aller de 10-15 à plus de 200 nucléotides de longueur. Leur présence de part et d'autre de la séquence intégrée signe l'implication d'un élément LINE-1 dans l'intégration (voir plus loin dans le texte).
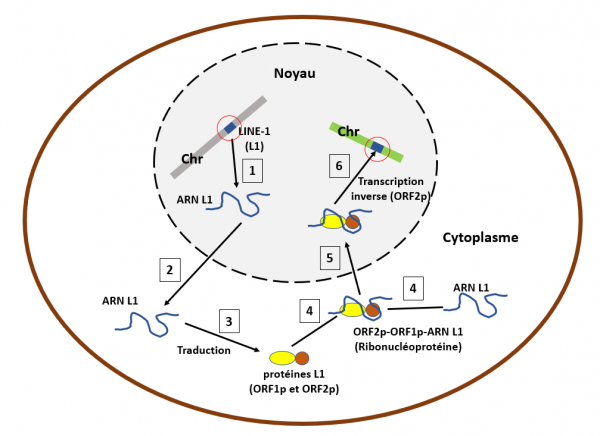
Schéma très simplifié du cycle de rétro-transposition d'un élément LINE-1
L'ADN codant pour un rétrotransposon LINE-1 est représenté par un rectangle bleu (cerclé en rouge) dans un chromosome (rectangle gris) dans le noyau de la cellule (ellipse colorée en gris clair). Etape 1 : l'ADN LINE-1 est transcrit en ARN messager (ARN L1). Etape 2: cet ARN bicistronique (codant pour deux protéines) est transporté dans le cytoplasme et traduit (étape 3) en deux protéines, ORF1p (reconnaissance de l'ARN L1) et ORF2p (endonucléase et transcriptase inverse). Etape 4 : ces protéines recrutent un ARN L1 et s'y associent pour constituer une ribonucléoprotéine. Etape 5 : cette ribonucléoprotéine pénètre dans le noyau cellulaire et après avoir reconnu une séquence spécifique dans un nouveau chromosome (rectangle vert), la protéine ORF-2p rétro-transcrit l'ARN L1 en ADN L1 (rectangle bleu cerclé de rouge) qu'elle insère dans ce chromosome. Les éléments LINE-1 se rétro-transposent ainsi dans le génome humain selon un processus "copier-coller".
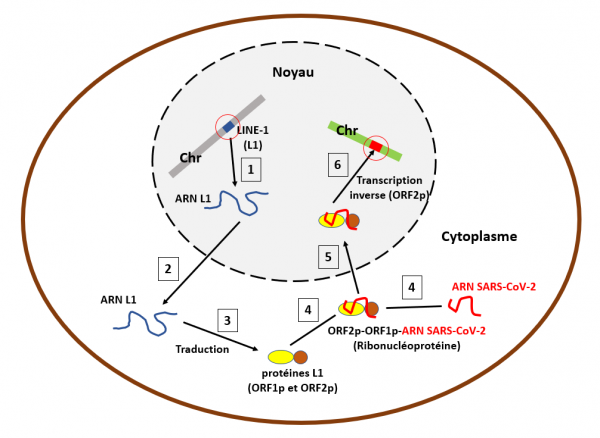
Schéma très simplifié de l'intégration du génome du SARS-CoV-2 dans le génome humain par un rétrotransposon LINE-1
L'ADN codant pour un rétrotransposon LINE-1 est représenté par un rectangle bleu (cerclé en rouge) dans un chromosome (rectangle gris) dans le noyau de la cellule (ellipse colorée en gris clair). Etape 1 : l'ADN LINE-1 est transcrit en ARN messager (ARN L1). Etape 2: cet ARN bicistronique (codant pour deux protéines) est transporté dans le cytoplasme et traduit (étape 3) en deux protéines, ORF1p (reconnaissance de l'ARN L1) et ORF2p (endonucléase et transcriptase inverse). Etape 4 : dans de rares cas (entre 1 et 5 sur 10000), ces protéines peuvent (bien que rarement) recruter un ARN différent de l'ARN L1, ici un ARN sous génomique du SARS-CoV-2 (ligne rouge). Elles s'y associent pour constituer une ribonucléoprotéine portant l'ARN SARS-CoV-2. Etape 5 : cette ribonucléoprotéine pénètre dans le noyau cellulaire et après avoir reconnu une séquence spécifique dans un nouveau chromosome (rectangle vert) la protéine ORF-2p rétro-transcrit l'ARN viral en ADN viral qu'elle insère dans ce chromosome (rectangle rouge cerclé de rouge). La cellule dans lequel cet évènement s'est produit est maintenant capable de produire de l'ARN SARS-CoV-2 même si elle n'est pas infectée par le virus.
Arguments en faveur de l'intégration du génome viral dans le génome de l'hôte
Articles de Zhang et al. BioRxiv2021; Zhang et al. PNAS 2021
Le 13 Décembre 2020, un groupe de chercheurs de l'Université de Harvard et du MIT publiait un premier article sur la plateforme BioRxiv (Zhang et al. BioRxiv 2020; article non examiné par des pairs) qui allait provoquer un grand nombre de réactions dans la communauté scientifique internationale. Ces chercheurs mettaient en évidence la possibilité que dans des cellules infectées, les ARN du SARS-CoV-2 puissent faire l'objet d'une transcription inverse en ADN puis d'une intégration dans le génome humain. Selon ces auteurs, la transcription des séquences intégrées permettrait d'expliquer les tests RT-PCR positifs chez les personnes guéries de l'infection. À l'appui de cette hypothèse, des ARN chimériques constitués de séquences virales fusionnées à des séquences cellulaires humaines étaient détectés dans des banques de données publiées de cellules cultivées infectées par le SARS-CoV-2 et de cellules primaires de malades infectés. Pour corroborer expérimentalement la possibilité d'une rétro-intégration virale, les chercheurs ont montré que les ARN du SARS-CoV-2 peuvent être transcrits de manière inverse dans des cellules humaines par la transcriptase inverse d'éléments LINE-1 ou par la transcriptase inverse du HIV-1, et que ces séquences d'ADN peuvent être intégrées dans le génome cellulaire et être ensuite transcrites en ARN chimérique homme-virus. L'étude a également révélé que l'expression des éléments LINE-1 est induite lors de l'infection par le SARS-CoV-2 ou par exposition aux cytokines dans des cellules en culture, ce qui suggère un mécanisme moléculaire qui pourrait sous-tendre la rétro-intégration du SARS-CoV-2 chez les malades de la COVID-19. Cette caractéristique de l'infection par le SARS-CoV-2 pourrait expliquer pourquoi certains patients peuvent continuer à produire de l'ARN viral après leur guérison.
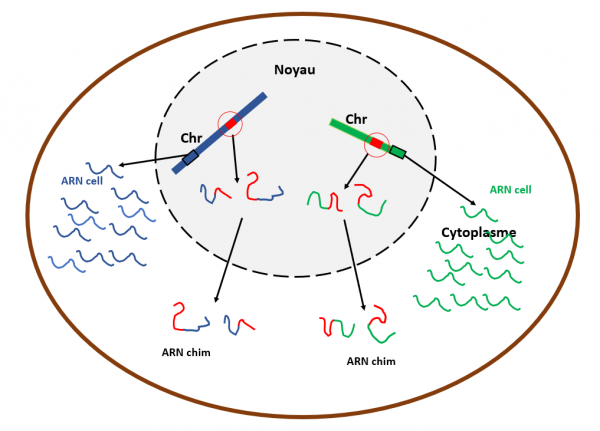
Schéma très simplifié sur la production d'ARN chimériques homme-virus après intégration du génome viral dans le génome de l'hôte
Dans la cellules schématisée ici, deux fragments du génome du SARS-CoV-2 (rectangles rouge cerclé de rouge) sont intégrés dans deux chromosomes différentes (rectangles bleu et vert). Dans un souci de simplification, seulement deux chromosomes sont représentés ici. Les gènes présents sur les deux chromosomes sont transcrits en ARN de couleur correspondante (ARN cell, lignes bleues et vertes). La transcription des fragments d'ADN viraux produit des ARN chimérique (ARN chim) du fait que l'ARN viral est associé à de l'ARN cellulaire adjacent comme montré ici, lignes rouge-bleu, bleu-rouge, rouge-vert et vert-rouge.
Ces mêmes chercheurs ont ensuite effectué des analyses supplémentaires, qu'ils ont publiées dans un deuxième article en mai dernier dans la revue de l'académie des sciences américaine (PNAS). Ces nouveaux travaux confirment les précédents et montrent que des copies d'ADN issues de séquences virales peuvent être intégrées au génome de cellules humaines infectées. Ces ADN chimériques homme-virus ont pu être isolés et séquencés. En particulier, des duplications des sites cibles flanquant les séquences virales et des séquences consensuelles de reconnaissance de l'endonucléase de LINE-1 sur les sites d'intégration y ont été retrouvées. Ce résultat est cohérent avec l'implication de rétrotransposons LINE-1 dans la formation des chimères. Les chercheurs ont également trouvé, dans certains tissus dérivés de patients, des éléments de preuve suggérant qu'une grande fraction de l'ADN du SARS-CoV-2 est transcrite à partir de copies d'ADN des séquences virales intégrées, générant des transcriptions chimériques homme-virus. Voir figure ci-dessous : scénario 1. Puisque seules des séquences sous-génomiques dérivées principalement de l'extrémité 3′ du génome viral (codant notamment pour la nucléocapside virale) sont généralement retrouvées, il est impossible qu'un virus SARS-CoV-2 infectieux puisse être produit dans ces conditions.
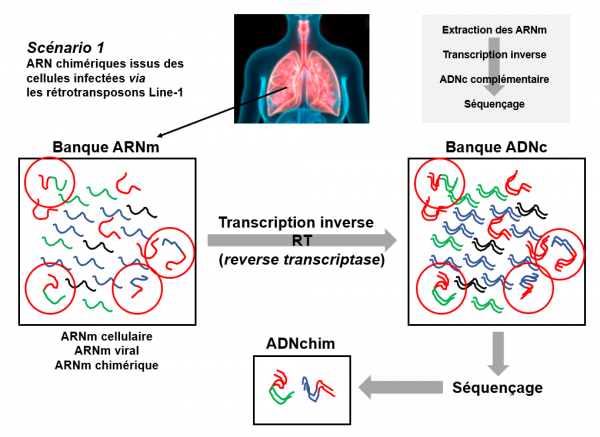
Scénario 1 : les ARN chimériques sont présents dans les cellules prélevées et analysées
Des cellules pulmonaires d'un malade infecté sont prélevées. Une banque d'ARN est constituée à partir de ce prélèvement. Parmi ces ARN on trouve de l'ARN cellulaire (traits bleu ou vert) et de l'ARN viral (traits rouge). Si le SARS-CoV-2 peut effectivement s'intégrer dans le génome des cellules de l'hôte, cette banque d'ARN contient également des ARN chimériques homme-virus (figurés par les traits rouge-vert, rouge-bleu). Afin d'être analysé par PCR, la banque d'ARN est convertie en banque d'ADNc par une transcription inverse à l'aide d'une rétrotranscriptase (RT). Les ARN (simple brin) sont ainsi convertis en ADN (double brin). Le séquençage permet de détecter des ADN chimériques homme-virus. Dans les cellules ces ADN chimériques seront transcrits en ARN chimérique homme-virus qui seront détectés par RT-PCR.
Article de Yin et al. Frontiers 2021
Dans cette étude, les chercheurs ont analysé le transcriptome (ensemble des ARN d'une cellule ou d'un tissu, voir plus loin, en détail) de cellules dérivées de poumons humains infectées par le MERS-CoV, le SARS-CoV et le SARS-CoV-2. Les résultats montrent que l'infection par ces coronavirus induit une augmentation de l'expression des rétrotransposons LINE-1 par stimulation épigénétique. La même observation a été faire dans des organoïdes intestinaux humains infectés par le SARS-CoV-2. Cette expression accrue des rétrotransposons peut conduire à une instabilité accrue du génome et à une augmentation anormale de l'expression de gènes cellulaires. De fait, les personnes ayant un niveau basal plus élevé de rétrotransposons, comme les patients cancéreux et les personnes âgées, pourraient présenter un risque accru d'infection symptomatique. De plus, les résultats apportent des preuves soutenant l'héritage épigénétique à long terme de la régulation positive des rétrotransposons. Des ARN chimériques de rétrotransposon et d'ARN du SARS-CoV-2 ont été observés, suggérant une invasion potentielle du génome humain par des fragments viraux, la partie avant (5') et arrière (3') du génome du SARS-CoV-2 se prêtant plus facilement à la formation d'ARN chimériques hôte-virus. En résumé, les auteurs proposent que le SARS-CoV-2 en se multipliant dans les cellules humaines interagit avec les rétrotransposons de type LINE-1, une situation pouvant provoquer des symptômes plus graves chez les patients atteints de maladies sous-jacentes.
Arguments contre l'intégration du génome viral dans le génome de l'hôte
La formation d'ARN chimériques homme-virus SARS-CoV-2 pourrait cependant avoir une autre origine que l'intégration du génome viral dans le génome de l'hôte.
Le transcriptome et le séquençage des ARN messagers (RNAseq)
Le transcriptome représente l'ensemble des ARN présents dans une cellule, un tissu ou un organe à un temps t. Le transcriptome est en perpétuelle évolution. En effet, la transcription des gènes en ARNm (messager) varie considérablement selon les conditions physiologiques : nutrition, jeûne, stress, influence de l'environnement, pathologies, etc. Le transcriptome constitue une donnée d'une extrême importance pour comprendre le fonctionnement de la cellule, du tissu ou de l'organe et éventuellement les pathologies dont il, ou elle, peut être atteint.
Le RNASeq (séquençage de l'ARN, "RNA sequencing" en anglais), également appelé "séquençage aléatoire du transcriptome entier" est une technologie basée sur le séquençage à haut débit pour caractériser et quantifier les ARN messagers issus de la transcription du génome. Cependant, le séquençage direct de l'ARN n'est pas possible. Seul l'ADN peut être séquencé. Pour connaître le transcriptome il faut donc passer par une étape ADN.
Pour cela l'ARN est isolé à partir de la cellule, du tissu ou de l'organe. En général on sélectionne les ARN messagers (ARNm, porteurs d'une séquence poly A en position 3') qui codent pour les protéines. Une fois isolés et purifiés les ARNm sont rétro-transcrits en ADN complémentaires (ADNc, copie exacte de l'ARNm, sans les séquences introniques) par une transcriptase inverse (RT, "reverse transcriptase" en anglais). Après amplification des ADNc par PCR on obtient une banque d'ADNc qui reflète l'ensemble des ARNm exprimés au moment de l'extraction de l'ARN. Le séquençage des ADNc permet alors d'évaluer l'abondance des ARNm relatifs à chaque gène, c'est-à-dire le nombre de fois qu'un même ADNc a été séquencé. Ainsi, il est possible de déterminer l'expression relative des gènes dans une condition physiologique donnée, en comparaison à une condition physiologique de référence.
Articles de Yan et al. J Virol 2021 et Yan et al. BioRxiv 2021
Ces chercheurs ont tenté de démontrer que dans le conditions naturelles, le SARS-CoV-2 ne peut intégrer son génome dans celui de l'hôte. Contrairement aux véritables événements biologiques tels que l'épissage de l'ARNm et les événements de réarrangement du génome qui génèrent des fragments de séquence chimériques reproductibles à travers différents isolats biologiques, les chercheurs ont constaté que dans plus de 100 banques de RNA-seq de patients atteints de COVID-19 et de lignées cellulaires infectées, les ARN chimériques hôte-virus n'étaient pas reproductibles d'une banque à une autre. En fait, les banques ARN-seq sont intrinsèquement sujettes à des erreurs pendant la transcription inverse de l'ARN en ADNc conduisant à la production d'ARN chimériques. Selon ces chercheurs, les ARN chimères homme-virus n'étaient pas présents dans le prélèvement de départ mais ont été créés artificiellement par la transcriptase inverse lors de la construction de la banque RNA-seq. Voir ci-dessous: scénario 2.
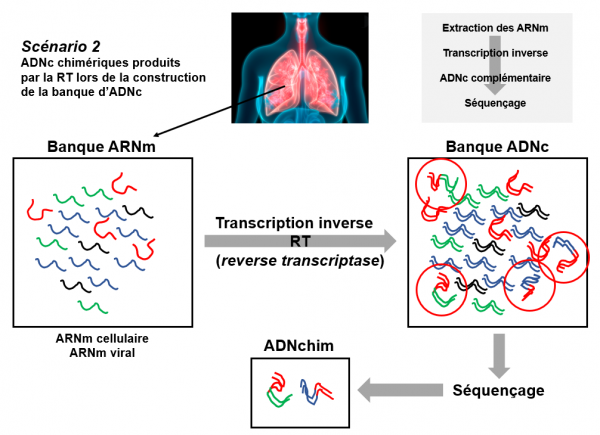
Scénario 2 : les ARN chimériques ne sont pas présents dans les cellules prélevées mais créés lors de la préparation de la banque d'ADNc
Des cellules pulmonaires d'un malade infecté sont prélevées. Une banque d'ARN est constituée à partir de ce prélèvement. Parmi ces ARN on trouve de l'ARN cellulaire (traits bleu ou vert) et de l'ARN viral (traits rouge). Afin d'être analysé par RT-PCR, la banque d'ARN est convertie en banque d'ADNc par une transcription inverse à l'aide d'une rétrotranscriptase (RT). Les ARN (simple brin) sont ainsi convertis en ADN (double brin). Au cours de cette préparation, des chimères homme-virus (traits rouge-bleu et rouge-vert) sont produits artificiellement. Le séquençage permet de détecter des ADN chimériques homme-virus qui ne représentent que des artéfacts expérimentaux et n'ont aucune signification biologique.
Les chercheurs ont pu mesurer ce phénomène de la façon suivante. Ils ont ajouté à de l'ARN humain un ARN témoin-test provenant d'une espèce non apparentée, la drosophile (mouche). Après construction de la banque ARN-seq avec ces ARN, ils ont trouvé que ~1 % des séquences ARN-seq étaient artificiellement chimériques homme-mouche. Dans l'ARN-seq du SARS-CoV2, ils ont constaté que la fréquence des événements chimériques n'était, en fait, pas supérieure à ce " bruit de fond" de 1%. Enfin, ils ont mis au point une nouvelle approche expérimentale pour enrichir les séquences SARS-CoV2 à partir de l'ARN total de cellules infectées. Alors que cette méthode a fortement enrichi les séquences virales, elle n'a pas enrichi les séquences d'ARN chimériques homme-virus. Ceci suggère que la majorité de ces événements sont, selon toute vraisemblance, des artefacts de la construction des banques. En conclusion, les résultats indiquent que les événements chimériques observés dans les banques de séquençage de l'ARN provenant de cellules infectées par le SARS-CoV2 sont au mieux extrêmement rares et résultent probablement d'artefacts expérimentaux liés à leur construction et non à l'intégration du génome viral dans le génome de l'hôte.
Articles de Kazachenka et al. BioRxiv 2021; Kazachenka et al. Front Microbiol 2021
Dans ces articles, les chercheurs ont également recherché l'origine des ARN chimériques homme-virus SARS-CoV-2 dans des banques RNA-seq. Des séquences chimériques ont été fréquemment détectées entre l'ARN du SARS-CoV-2 et l'ARN transcrit à partir de l'ADN mitochondrial ou de l'ADN adénoviral présent dans les lignées cellulaires transfectées par un adénovirus surexprimant ACE2, ce qui ne pouvait être le résultat de l'intégration du SARS-CoV-2. Pour l'heure, aucune rétrotranscription d'un ARN viral dans l'ADN mitochondrial n'a été observée. En outre, des ARN chimériques entre des ARN nucléaires et l'ARN du SARS-CoV-2 impliquaient le plus souvent les mêmes gènes hôtes très fortement exprimés et étaient fortement enrichies en séquences exoniques de l'hôte, plutôt qu'introniques ou intergéniques, largement majoritaires dans le génome. Un séquençage global du génome (whole genome sequencing) pourrait permettre de détecter des séquences virales du SARS-CoV-2 intégrées au génome humain, mais ces données ne sont pour le moment pas disponibles. Les auteurs concluent que les ARN chimériques homme-virus SARS-CoV-2 trouvées dans les banques RNA-seq peuvent être générés pendant la préparation de la banque et non via une intégration du génome viral dans le génome des cellules hôtes humaines.
Article de Smits et al. BioRxiv 2021
Dans cet article, les auteurs ont utilisé la même méthode de séquençage de l'ADN que Zhang et al. (voir plus haut) sur les mêmes cellules HEK293T infectées par le SARS-CoV-2, en absence de surexpression de LINE-1, et n'ont trouvé aucune preuve de l'existence de séquences virales sous forme d'ADN. Les auteurs concluent que si le phénomène de rétro transcription d'ARN du SARS-CoV-2 par LINE-1 est plausible au plan du mécanisme, il est probablement très rare, comme c'est le cas pour d'autres ARN messagers.
Articles Kazachenka Front. Microbiol. 2021 et Gaebler et al. Nature 2021
Les travaux décrits dans ces deux publications montrent que longtemps après guérison il existe des "réservoirs" au sein desquels le virus est capable de survivre et se répliquer. Ainsi on peut détecter de l'ARN viral à distance de la guérison sans que cela nécessite une intégration de son génome chez l'hôte.
Articles de Elfman et al. Stem Cell Int 2018 et Shi et al. Adv Clin Chem 2021
A côté des possibilités de formation de chimères homme-virus résultant de la construction des banques ARN-seq, discutées ci-dessus, un autre argument peut être invoqué contre la formation naturelle de chimères homme-virus médiée par les éléments LINE-1.
Divers travaux résumés dans les deux publications citées ci-dessus montrent que la formation d'ARN chimériques dans les cellules est un évènement relativement courant. Les technologies de séquençage de nouvelle génération (notamment RNA-seq) ont permis l'identification d'un grand nombre d'ARN chimériques. Plusieurs mécanismes ont été décrits pour expliquer la formation de telles chimères comme par exemple la fusion de gènes lors de réarrangements chromosomiques, un évènement fréquemment invoqué dans l'étiologie de cancers. Mais deux autres mécanismes permettant la génération d'ARN chimériques existent. Ce sont le trans-épissage et le cis-épissage de gènes adjacents (cis-SAGe). En bref, lors de la transcription d'un gène, un ARN pré-messager est produit et ce pré-ARNm doit être épissé dans le noyaux pour éliminer les séquences introniques non codantes afin d'aboutir à l'ARNm (messager) qui sera traduit en protéine. Le phénomène d'épissage est complexe et peut donner lieu à des recombinaisons entre ARNm issus de différents gènes, conduisant à la formation d'ARN chimériques. Contrairement à ce que l'on pensait il y a encore quelques années, ces ARN chimériques ne sont pas propres aux cellules cancéreuses, mais sont également présents dans les cellules et tissus normaux. Ainsi Singh et al. (Nucl Acid Res 2020) ont mis en évidence des milliers de telles chimères dans divers tissus normaux, démontrant l'ubiquité de ces ARN dans la physiologie normale.
MAIS, le SARS-CoV-2 n'a pas d'étape nucléaire dans son cycle de vie. Ces mécanismes ne peuvent donc pas être responsables a priori des chimères homme-virus.
Conclusion
Il n'y a donc pour l'heure pas encore de consensus scientifique clair sur la proposition selon laquelle le génome du SARS-CoV-2 pourrait s'intégrer dans le génome humain. En d'autres termes les scénarii 1 et 2 présentés ci-dessus ne peuvent être départagés. Il est cependant important de rappeler que des cas d'intégration de génome de différents virus à ARN (Bornavirus, Ebola virus, Marburg virus, virus de la stomatite vésiculaire, virus de la chorioméningite lymphocytaire) par des élément LINE-1 ont déjà été observés, suggérant ainsi que le mécanisme d'intégration du SARS-CoV-2 discuté ici est plausible, sinon certain. Par contre, il semble que l'infection par le SARS-CoV-2 entraîne une augmentation de l'expression des éléments LINE-1, soit directement, soit indirectement via le stress oxydatif engendré par le virus. Une telle situation pourrait avoir des conséquences néfastes sur les malades du fait de l'instabilité génétique qu'elle engendre.
A suivre donc…
Pourquoi est-il hautement improbable que les vaccins à ARN conduisent à une intégration de l'ADN complémentaire codant pour la protéine S dans le génome humain ?
Les antivaccins se sont rapidement emparés des résultats sur la possibilité d'une intégration du génome viral dans le génome de l'hôte afin de propager l'idée FAUSSE selon laquelle les vaccins à ARN pourraient conduire à une intégration génomique de l'ARN codant pour la protéine S du SARS-CoV-2.
Une telle intégration, si elle était possible, pourrait en effet conduire à des effets néfastes et bien connus tels que des modifications de l'expression de gènes divers, incluant notamment les oncogènes ou les gènes anti-cancérogènes (silencers), ou des modifications chromosomiques. Cette intégration pourrait aussi avoir des effets bénéfiques, comme par exemple le maintien d'une immunité humorale et cellulaire contre ce virus.
Dans les cellules infectées par le SARS-CoV-2, les ARN viraux sont continuellement synthétisés du fait de la multiplication active des virions et restent présents pour de longues périodes, entre plusieurs jours, jusqu'à plusieurs mois. L'ARN viral majoritairement suspecté d'intégration au sein du génome des cellules infectées est celui codant pour la nucléocapside (NC) du SARS-CoV-2. En effet, cet ARN est très largement majoritaire parmi tous les ARN sous-génomiques produits par le virus, la nucléocapside qui sert à empaqueter le génome viral dans l'enveloppe du virus étant elle-même largement majoritaire parmi l'ensemble des protéines produites par le virus. D'où la probabilité maximale de retrouver cet ARN codant pour la nucléocapside intégré dans le génome de l'hôte, par rapport aux autres ARN sous-génomiques viraux et, en particulier, celui codant pour la protéine S. En tout état de cause, les travaux décrits ici suggèrent que si cet évènement existe, il est extrêmement rare.
En revanche, dans les deux vaccins à ARN Pfizer et Moderna couramment utilisés aujourd'hui, l'ARN codant pour la protéine S du SARS-CoV-2 est administré en intramusculaire en deux doses de 30 ou 100 microgrammes, respectivement, au sein de nanoparticules lipidiques dont l'unique but est de le protéger, l'ARN étant une molécule extrêmement fragile. Le nombre des cellules "touchées" par le vaccin est très faible et leur temps d'exposition au vaccin très court comparé à la durée d'une infection (jours-semaines-mois). De plus, l'ARN du vaccin, injecté en petite quantité, n'est pas reproduit dans les cellules mais rapidement dégradé dès que sa traduction en protéine S est terminée. Une étude portant sur 50 gènes différents a montré que 80% des ARN messagers survivent moins de 2 minutes après avoir été transcrits (Becskei et al. Science Daily, 2017). La probabilité que l'ARN vaccinal codant pour la protéine S soit retro-transcrit et intégré au génome des cellules infectées est donc extrêmement faible, pour ne pas dire nulle, ce que l'objectivité scientifique m'interdit de dire.
En tout état de cause, le danger potentiel que représente l'intégration d'un génome étranger/viral dans le génome humain serait plus important chez le sujet infecté que chez le sujet vacciné. Enfin, il a été démontré que ce phénomène d'intégration génomique est possible pour d'autres virus.
Bibliographie
- Beauregard et al. The take and give between retrotransposable elements and their hosts. Ann. Rev. Genet. 2008
- Beck et al. LINE-1 Elements in Structural Variation and Disease. Ann. Rev. Genomics Hum. Genet. 2011
- Belyi et al. Unexpected Inheritance: Multiple Integrations of Ancient Bornavirus and Ebolavirus/Marburgvirus Sequences in Vertebrate Genomes. Plos Pathogens 2010
- Boissinot et al. The Evolution of LINE-1 in Vertebrates. Genome Biol. Evol. 2016
- Elfman et al. Chimeric RNA in Cancer and Stem Cell Differentiation. Stem Cell Int. 2018
- Feng et al. Human L1 Retrotransposon Encodes a Conserved Endonuclease Required for Retrotransposition. Cell 1996
- Gaebler et al. Evolution of antibody immunity to SARS-CoV-2. Nature 2021
- Horie et al. Endogenous non-retroviral RNA virus elements in mammalian genomes. Nature 2010
- Horie et al. Non-Retroviral Fossils in Vertebrate Genomes. Viruses 2011
- Huang et al. Recurrence of SARS-CoV-2 PCR positivity in COVID-19 patients: a single center experience and potential implications. MedRxiv 2020
- Kazachenka et al. SARS-CoV-2-host chimeric RNA-sequencing reads do not necessarily signify virus integration into the host DNA. BioRxiv 2021
- Kazachenka et al. SARS-CoV-2-Host Chimeric RNA-Sequencing Reads Do Not Necessarily Arise From Virus Integration Into the Host DNA. Front. Microbiol. 2021
- Kazazian et al. The impact of L 1 retrotransposons on the human genome. Nature 1998
- Lee et al. Somatic APP gene recombination in Alzheimer’s disease and normal neurons. Nature 2018
- Mahmood et al. Aging‑associated distinctive DNA methylation changes of LINE‑1 retrotransposons in pure cell‑free DNA from human blood. Sci. Rep. 2020
- Moran et al. High Frequency Retrotransposition in Cultured Mammalian Cells. Cell 1996
- Rodic et al. Long Interspersed Element–1 (LINE-1): Passenger or Driver in Human Neoplasms ? Plos Genetics 2013
- Shi et al. Chimeric RNAs in cancer. Adv. Clin. Chem. 2021
- Smits et al. Human genome integration of SARS-CoV-2 contradicted by long-read sequencing. BioRxiv 2021
- Szak et al. Molecular archeology of L1 insertions in the human genome. Genome Biol. 2002
- Szak et al. Identifying related L1 retrotransposons by analyzing 3' transduced sequences. Genome Biol. 2003
- Viollet et al. L1 retrotransposition. The snap-velcro model and its consequences. Mobile Gen. Elem. 2014
- Wei et al. Human L1 Retrotransposition: cis-Preference versus trans-Complementation. Mol. Cell Biol. 2001
- Yan et al. Host-virus chimeric events in SARS-CoV2 infected cells are infrequent and artifactual. BioRxiv 2021
- Yan et al. Host-virus chimeric events in SARS-CoV2 infected cells are infrequent and artifactual. J. Virol. 2021
- Yin et al. Exogenous Coronavirus Interacts With Endogenous Retrotransposon in Human Cells. Front. Cell. Infect. Microbiol. 2021
- Yuan et al. Recurrence of positive SARS‑CoV‑2 viral RNA in recovered COVID‑19 patients during medical isolation observation. Sci. Rep. 2020
- Zhang et al. SARS-CoV-2 RNA reverse-transcribed and integrated into the human genomeBioRxiv 2020
- Zhang et al. Reverse-transcribed SARS-CoV-2 RNA can integrate into the genome of cultured human cells and can be expressed in patient-derived tissues. Proc. Natl. Acad. Sci. 2021
Date de dernière mise à jour : 01/08/2024
Ajouter un commentaire