Ribosomes : un nouvel étage de la régulation génique
Dans le cadre du dogme ADN - ARN - protéines, le ribosome est la machine biologique qui dans le cytosol des cellules déchiffre le code génétique contenu dans les ARN messagers et le traduit en protéines correspondantes.
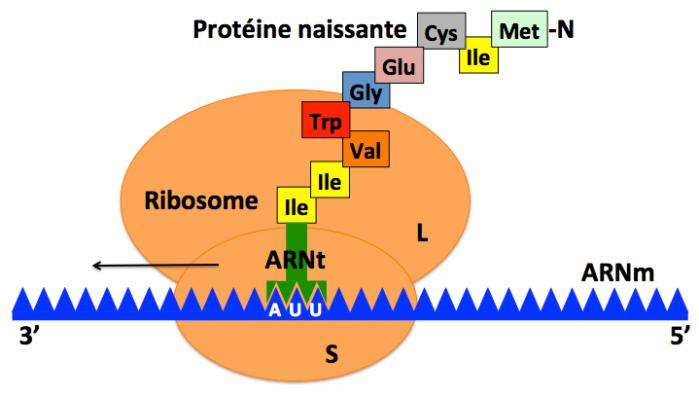
Schéma très simplifié du ribosome. Chaque ribosome est constitué de deux sous-unités, la grande (L, large) et la petite (S, small). Chez les eukaryotes, la petite sous-unité (40S) est constituée d’un ARN ribosomique (ARNr, 18S) de 1900 nucléotides et de 33 protéines ribosomiques ; la grande sous-unité est constituée de trois ARNr (5S, 28S et 5,8S) de 120, 4700 et 160 nucléotides et de 46 protéines ribosomiques (non détailés sur la figure). La sous-unité S se lie sur l’ARN messager (ARNm) pour en assurer la traduction dans le cytosol. Elle recrute ensuite les ARNs de transfer (ARNt) qui fixent spécifiquement un acide aminé (isoleucine dans l’exmple) et viennent s’hybrider à l’ARNm par appariement des bases nucléotidiques, puis la sous-unité L. Sur la figure, l’exemple de l’ARNt de l’isoleucine (en vert ; qui s’hybride au codon AUU qui code pour l’isoleucine, Ile) est représenté. La sous-unité L assure la liaison entre les acides aminés convoyés par les ARNt et la chaîne polypeptidique (protéine) naissante. La flèche noire indique le sens de déplacement du ribosome sur l’ARNm.
On pensait jusqu’ici que le rôle du ribosome se limitait à une traduction précise mais passive de la séquence des ARNm en protéines qui constituent l’ensemble de nos organes, tissus et systèmes. On pensait également que les 1 à 10 millions de ribosomes que compte chaque cellule étaient interchangeables.
Et bien non !
Certes, le ribosome reste l’acteur majeur de la traduction, mais pas seulement. C’est ce que montrent deux articles publiés cet été par l’équipe de Maria Barna de l’Université de Stanford (Simsek et al. Cell 2017 ; Shi et al. Molecular Cell 2017).
Des études précédentes, notamment certaines issues de l’équipe de Maria Barna (Kondrasov et al. Cell 2011) suggèraient que des protéines associées aux ribosomes pouvaient avoir un impact sur le contrôle et la fidélité de la traduction des protéines. L’hypothèse selon laquelle des ribosomes “spécialisés” possédant des activités fonctionnelles “uniques” pourraient moduler l’expression de gènes, ou réguler l’homéostasie de cellules souches durant le développement avait été formulée. Mais la “spécialisation” de l’activité traductionnelle des ribosomes au niveau de leurs constituants moléculaires n’avait pas été démontrée.
C’est chose faite.
Une première partie du travail (Simsek et al. Cell 2017) avait pour objectif d’identifier l’ensemble des protéines associées aux ribosomes. Isoler les ribosomes du reste des protéines présentes dans le cytosol n’est pas chose facile. Pour résoudre ce problème, les chercheurs ont marqué les deux sous-unités des ribosomes de cellules embryonnaires de souris avec une “étiquette” FLAG (un octapeptide) sur deux protéines ribosomiques de surface, eL36 (sous-unité L) et eS17 (sous-unité S) par la technologie CRISPR/Cas9 (voir sur ce site). L’étiquette FLAG a ainsi permis d’isoler spécifiquement les ribosomes par immunoaffinité et immunoprécipitation avec des anticorps anti-FLAG. Ces ribosomes purifiés ont été analysés par spectroscopie de masse couplée à des analyses de bioinformatique des interactions protéine-protéine et d’ontologie génique. Les chercheurs ont ainsi pu identifier et quantifier les protéines associées aux ribosomes (RAP, Ribosome Associated Proteins). Celles-ci sont au nombre d’environ 450 !!! Parmi les RAPs, seulement 15% et 3% sont spécifiquement et respectivement liées aux ARNm et aux peptides naissants mais pas au ribosome lui-même. Le reste des RAPs (soient environ 430 protéines) incluant les 80 protéines ribosomiques classiques constituent donc ce que les chercheurs ont appelé le ribo-interactome.
Les protéines classiquement connues pour leur rôle dans la traduction et le contrôle du repliement et de la struture protéique ont bien évidemment été retrouvées dans les protéines associées aux ribosomes. Cependant et curieusement, les analyses ont fait apparaître d’autres RAPs impliquées dans des fonctions telles que le métabolisme et le cycle cellulaire ainsi que dans le processing des ARNm, comme des hélicases et des protéines impliquées dans le transport, la maturation (splicing) et l’inhibition de l’expression des gènes par les microRNAs.
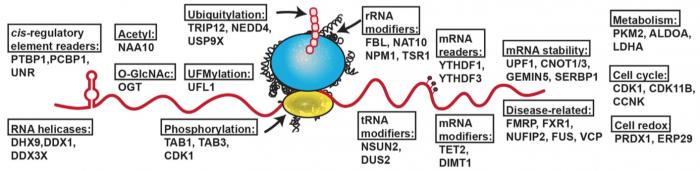
Schéma du ribo-interactome. Ici encore il s'agit d'une vue simplifiée des protéines associées aux ribosomes. Au centre les deux sous-unités (L en bleu et S en jaune). La ligne en rouge figure l'ARNm. Les divers cadres font référence au type de fonction des protéines présentes dans l'interactome. Au dessous des cadres quelques exemples de gènes codant pour ces protéines. (Figure 3B de l'article Simsek et el. Cell 2017)
Ces résultats montrent donc qu’il existerait une interconnection entre le ribosome et d’importants processus cellulaires.
Dans une deuxième partie de ce travail (Shi et al. Molecular Cell 2017) les chercheurs ont examiné l’hétérogénéité ribosomique non plus au niveau des protéines associées aux ribosomes (les RAPs) mais au niveau des protéines ribosomiques dans des cellules embryonnaires de souris.
En utilisant les mêmes méthodes d’analyse et de quantification des protéines ribosomiques (RP) que celles utilisées précédemment, les chercheurs ont montré que les ribosomes en cours de synthèse protéique sont fortement hétérogènes dans leur composition en RP, et que des groupes d’ARNm qui codent pour des agents régulateurs de fonctions biologiques telles que le métabolisme, la prolifération et la survie cellulaires sont préférentiellement et différentiellement traduits par des types spécifiques de ribosomes.
En résumé, les ribosomes ne sont pas tous égaux !!!
Il semblerait donc que dans un type cellulaire unique (ici les cellules souches embryonnaires), d’une part les ribosomes sont fortement hétérogènes dans leur composition en protéines ribosomiques (RP) et en protéines ribosomiques associées (RAP), et que d’autre part cette hétérogénéité a un impact décisif sur la nature des ARNm traduits par ces ribosomes dans la cellule.
A un type de ribosome pourrait correspondre une batterie d’ARNm spécifiques.
Il s’agit donc là d’un nouvel étage de la régulation de l’expression des gènes.
Ces travaux amènent plusieurs questions :
- comment les ribosomes sont-ils capables de “choisir” les ARNm qu’ils vont traduire préférentiellement ?
- l’hétérogénéité de composition des ribosomes varie-t-elle d’un type cellulaire à l’autre ?
- l’hétérogénéité de composition des ribosomes varie-t-elle d’un état de la cellule à l’autre durant la différenciation, la prolifération, la senescence, l’apoptose, etc. ?
A suivre…
Date de dernière mise à jour : 20/09/2017
Ajouter un commentaire
