Tests génétiques, antigéniques et sérologiques
Résumé tout public
Depuis le début de la pandémie de COVID-19 on parle beaucoup des tests, et pour cause, ils revêtent une importance fondamentale dans le suivi de la pandémie. Mais qu’en est-il exactement et que peut-on leur faire dire ?
Il existe schématiquement trois types de tests:
-
les tests RT-PCR (transcription inverse et réaction en chaîne par polymérase) et RT-LAMP (amplification isothermique sans extraction d’ARN) sont basés sur l'amplification du génome viral. Ces tests sont pratiqués sur les prélèvements nasopharyngés, salivaires, selles, urines, etc., et permettent de détecter la présence de l'ARN du virus. La positivité de ces tests signifie que la personne est infectée mais pas forcément malade. En effet, les porteurs asymptomatiques sont positifs alors qu'ils ne sont pas malades. De plus, ces tests ne renseignent pas sur la nature de l'ARN viral détecté qui peut être soit issu du virus vivant, soit être un simple reliquat du génome viral détruit par le système immunitaire. Au plan technique, le test RT-LAMP est plus facile à réaliser que le test RT-PCR avec des temps d'analyse et de lecture des résultats beaucoup plus courts. Il est réalisé sur tous les types de prélèvements mentionnés ci-dessus, mais est principalement destiné à détecter le génome viral dans la salive, dans le but d'éviter le prélèvement nasopharyngé parfois douloureux pour les patients. Un test rapide EasyCoV développé par un consortium Montpellièrain et utilisant la technologie RT-LAMP sur prélèvement salivaire, vient d'être autorisé par la Haute Autorité de Santé (HAS) sur les malades symptomatiques uniquement.
-
Le test RT-PCR réalisé sur un prélèvement nasopharyngé reste (pour le moment) le test de référence.
- les tests antigéniques sont basés sur la détection d'une ou plusieurs protéines virales (les antigènes). Ils sont pratiqués sur les mêmes prélèvements que ceux mentionnés ci-dessus. Ils sont bien moins sensibles que les tests RT-PCR et LAMP, mais ils peuvent être réalisés très facilement et rapidement grâce à des dispositifs de tests rapides (15 à 30 minutes) ne nécessitant pas de technologie complexe et pouvant être réalisés en pharmacie ou "à la maison". Ils peuvent être utilisés pour diagnostiquer une infection à SARS-CoV-2 quand les tests RT-PCR ne sont pas disponibles ou que l’utilité clinique du dépistage serait compromise par des délais trop longs d’obtention des résultats. Les tests antigéniques ne sont pas recommandés chez les cas asymptomatiques sauf chez les cas-contacts d’un cas confirmé. Par ailleurs, l’OMS ne recommande pas l’utilisation des tests rapides antigéniques pour éviter les faux-positifs. Ainsi, les tests antigéniques ne sont pas recommandés comme outils de surveillance en conditions de faible circulation du virus. Dans ce cas-là, le test par RT-PCR reste le test de référence.
- les tests sérologiques sont basés sur la détection d'anticorps anti-SARS-CoV-2 dans le sang du patient. Ces tests permettent de savoir si la personne est ou a été infectée par le virus. En effet, si tel est le cas elle a développé des anticorps anti-SARS-CoV-2. Comme dans le cas des tests antigéniques, de nombreux tests sérologiques rapides (15 à 30 minutes) ne nécessitant pas de technologie complexe et pouvant être réalisés en pharmacie ou "à la maison". Selon la HAS, ces tests sont remboursés y compris lorsqu’ils sont réalisés en médecine de ville depuis le 7 mars 2020. Cependant et en cas de résultat positif le test de sérologie rapide devra toujours être confirmé par un test sérologique réalisé en laboratoire de biologie médicale.
Enfin, la HAS rappelle que ces indications sont susceptibles d’être revues en fonction de l’évolution des connaissances scientifiques.
Pour ceux qui veulent en savoir davantage, la mise au point de ces tests et la façon dont ils fonctionnent sont décrites en détails dans l'article ci-dessous.
Avertissement
Il existe un très grand nombre de tests RT-PCR et RT-LAMP ciblant des séquences différentes du génome viral et utilisant des conditions expérimentales de températures et de durée des cycles d'amplification différents. De même il existe un très grand nombre de tests antigéniques et sérologiques qui utilisent des antigènes (protéines virales différentes) et des anticorps différents, ainis que des configurations différentes (ELISA ou tests en barrettes). Pour des raisons évidentes, j'ai dû me limiter à seulement un exemple de chacun de ces tests. Cependant, les méthodologies décrites ici restent généralement les mêmes quels que soient les tests utilisés.
Les tests RT-PCR
La technique de PCR est utilisée dans tous les laboratoires de recherche biomédicale depuis plus de trente ans et dans diverses domaines : isolement de fragments d'ADN, quantification d'ADN, séquençage de l'ADN, détection de mutations, expression génique, identification d'agents pathogènes, identification d'espèces animales, etc. Cette technique est également utilisée en médecine légale notamment pour l'identification de personnes (empreintes génétiques).
Dans le cadre de la pandémie de COVID-19, ce test permet de détecter la présence du génome viral (c’est à dire l’ARN du SARS-CoV-2) dans le prélèvement (nasopharyngé, buccal, salive, sang, selles, eaux usées, etc.).
Premier point, RT-PCR est une abréviation anglaise.
RT (reverse transcription) signifie en français : transcription inverse
PCR (polymerase chain reaction) signifie en français : réaction en chaîne par polymérase.
Cela signifie que ce test comporte deux étapes, une première étape de transcription inverse destinée à produire de l’ADN à partir de l’ARN viral, puis seconde une étape de réaction en chaîne qui consiste à amplifier la quantité d’ADN par utilisation d’une polymérase. Une fois suffisamment amplifié l’ADN peut être détecté et quantifié.
Le génome du SARS-CoV-2
Le SARS-CoV-2, responsable de la COVID-19, est un virus enveloppé dont le génome est un ARN simple brin d’une longueur approximative de 30 000 nucléotides. Ce génome code pour trois types de protéines virales, 4 protéines structurales (qui entrent dans la composition des virions, Enveloppe, Spike, Membrane et Nucléocapside), 16 protéines non structurales (NSP1 à NSP16) qui participent principalement à la réplication du virus dans les cellules, à sa production et à la façon dont il va influencer la réponse immunitaire de l’hôte et enfin, entre 6 et 8 protéines dites « accessoires » (3a, 6, 7a, 7b, 8 et 10) dont la fonction n’est pas encore clairement établie. Ce génome est représenté schématiquement ci-dessous.
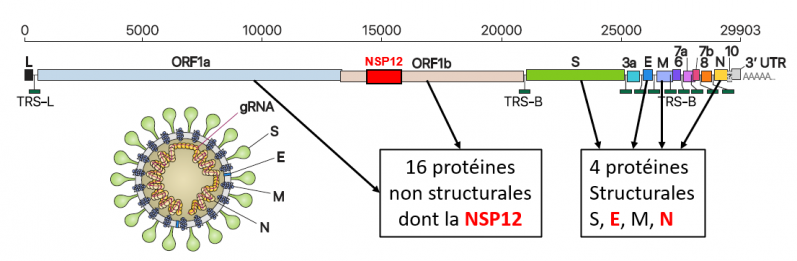
Génome du SARS-CoV-2
Longueur 29903 nucléotides comme indiqué sur la ligne supérieure. L et TRS-L et TRS-B : séquences régulatrices impliquées dans la réplication du génome viral. ORF1a et ORF1b (Open Reading Frame en anglais ; Cadre Ouvert de Lecture en français) désignent une large partie du génome qui code pour les protéines non structurales, dont notamment la NSP12. Partie basse à gauche : schéma du virion avec les protéines structurales S, E, M, et N. Le génome viral (ARN, gRNA) est empaqueté dans un « manchon » constitué de nombreuses molécules de protéine N. Les séquences les plus souvent ciblées par la PCR sont indiquées en rouge. (Schéma adapté de Kim et al. Cell 2020).
Transcription inverse
La rétro-transcriptase (RT) est une enzyme qui transcrit l’information génétique de l’ARN en ADN (cet ADN est alors appelé : ADNc, ADN complémentaire). Cette enzyme est utilisée par les rétrovirus (par exemple le virus du SIDA) et les systèmes rétrotransposons dans le génome. Le terme « rétro » signifie que cette enzyme agit à contre-courant du dogme de la biologie moléculaire. Ce dogme est : ADN à ARN à protéine. En d’autres termes, la RT viole le dogme de la biologie moléculaire en produisant de l’ADN à partir de l’ARN. Mesurer avec précision une concentration d’ARN est difficile en partie du fait de son instabilité. C’est la raison pour laquelle l’ARN est converti en ADN, la concentration de l’ADN résultant pouvant alors être amplifiée et mesurée avec précision par PCR.
Le SARS-CoV-2 est un virus à ARN. Quantifier ce virus dans les prélèvements cliniques revient donc à être capable de mesurer la concentration de son ARN dans ces prélèvements. D’où l’utilisation de la RT afin de synthétiser l’ADN complémentaire qui sera quantifié avec précision par PCR.
PCR
La PCR est une réaction en chaîne qui consiste à amplifier très fortement une séquence spécifique d’ADN par utilisation d’une ADN polymérase, une enzyme qui synthétise l’ADN, et de deux amorces sens et antisens, nécessaires d’une part à la sélection de la séquence à amplifier et d’autre au fonctionnement de la polymérase. Dans le cas du SARS-CoV-2 qui nous intéresse ici, les séquences spécifiques du génome viral ciblées par l’analyse en PCR sont celles codant pour la nucléocapside N (protéine fixant l’ARN viral au cœur des virions), la protéine d’enveloppe E, et la NSP12 (ARN polymérase dépendante d’ARN, la protéine qui synthétise l’ARN des virions). Une fois suffisamment amplifiée, la séquence d’ADN peut être détectée et quantifiée. Voir le schéma ci-dessous.
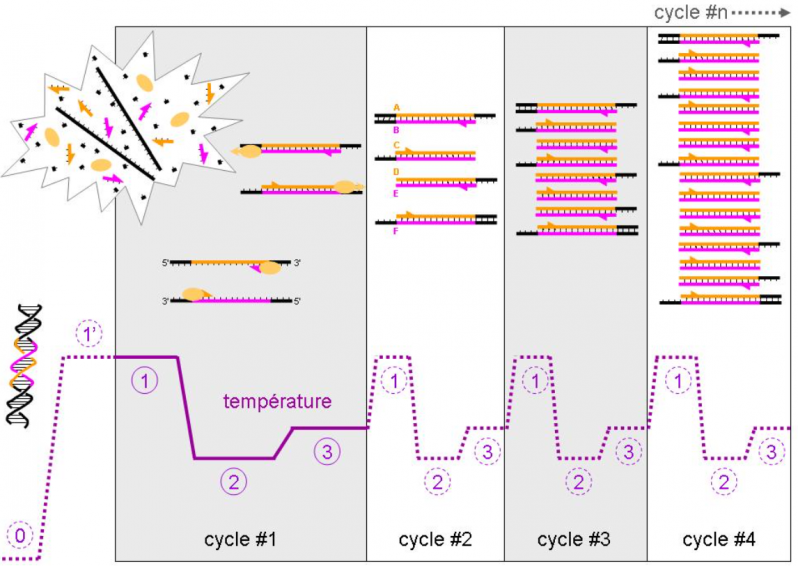
Description d’une réaction en chaîne par polymérase
Etoile en haut à gauche : les divers ingrédients du mélange utilisé. Barres noires : ADN un brin sens, un brin antisens. Demi-flèches en fushia : petit fragment d’ADN qui va servir d’amorce à la réaction de polymérisation du brin sens. Demi-flèches en orange : petit fragment d’ADN qui va servir d’amorce à la réaction de polymérisation du brin antisens. Ces deux fragments (appelés amorces sens et amorce antisens) ont été synthétisés en laboratoire pour encadrer la région d’ADN que l’on souhaite amplifier. Cette région, appelée amplicon, est matérialisée en couleurs (fushia et orange) sur le schéma de l’ADN en double hélice (double brin) à gauche de la figure. Les ellipses en jaune figurent la polymérase (enzyme qui va synthétiser l’ADN). Les petits blocs noir figurent les nucléotides (constituants de l’ADN, Adénine, Thymine, Guanine et Cytosine) nécessaires à la synthèse de l’ADN.
Cycle # 1. Etape 1, dite de dénaturation. Montée en température jusqu’à 95°C pendant environ 1 minute, afin de séparer les deux brins de la double hélice d’ADN. Rappel, à température normale (37°C, ou inférieure) les deux brins de l’ADN s’associent par appariement entre les bases A-T et G-C pour former une double hélice. A 95°C aucun appariement des brins d’ADN ne peut se produire.
Etape # 2, dite d’hybridation des amorces. Descente en température jusqu’à environ 55 à 65°C pendant une durée qui peut être comprise entre quelques secondes et 1 minute selon la longueur de l’amplicon. Pendant cette étape les amorces sens et antisens s’hybrident par appariement A-T et G-C avec les deux brins de l’ADN. A noter que ces conditions de température sont plus favorables à l’hybridation amorce-ADN qu’à l’hybridation brin sens-brin antisens de l’ADN (beaucoup plus longs).
Etape # 3, dite d’élongation. Remontée en température jusqu’à environ 68 à 72°C pendant une période comprise entre quelques secondes et 3 minutes selon la longueur de l’amplicon. Pendant cette étape la polymérase synthétise les brins sens et antisens de l’amplicon en se fixant sur les hybrides amorce-ADN.
Cycle # 2. Les mêmes étapes sont répétées. La quantité d’ADN synthétisé double (en théorie).
Cycle # 3. Les mêmes étapes sont répétées. La quantité d’ADN synthétisé double encore (en théorie).
Cycle # 4. Les mêmes étapes sont répétées. La quantité d’ADN synthétisé double encore (en théorie).
En général le nombre maximum de cycles est compris entre 40 et 45. (Source Wikipedia)
L’accumulation de l’ADN est visualisée par augmentation de fluorescence. Pour cela un fluorochrome (par exemple le SYBR Green, un agent intercalant) est ajouté dans le milieu réactionnel. Cette molécule ne produit pas de fluorescence à l’état libre. Cependant elle a une forte affinité pour l’ADN double brin et c’est uniquement lorsqu’elle est fixée sur l’ADN qu’elle devient fluorescente. L’enregistrement de la fluorescence au cours des divers cycles de PCR permet donc de visualiser la synthèse de l’ADN en temps réel (voir figure ci-dessous).
La réaction d’amplification de l’ADN par PCR est réalisée dans un appareil appelé thermocycleur. Cet appareil couplé à un ordinateur est programmé par l’expérimentateur afin de produire les cycles de PCR, nombre maximum, durée de chaque cycle, montées et descentes en température, analyse en temps réel et enregistrement des courbes de fluorescence. Un exemple des résultats obtenus est représenté ci-dessous.
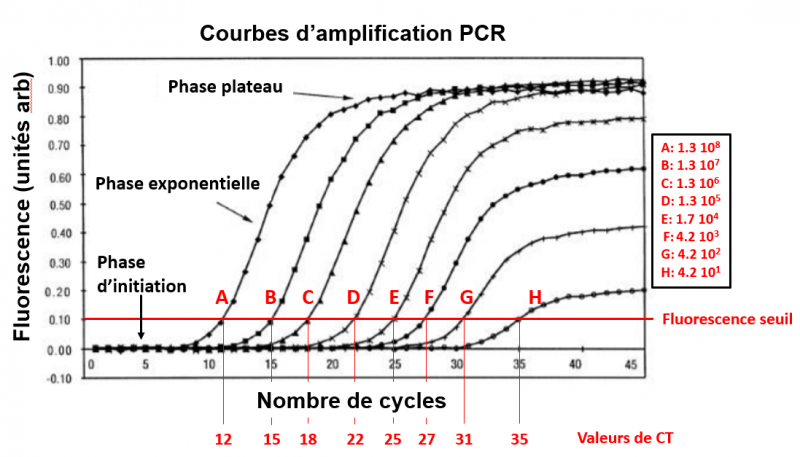
Courbes d’amplification de l’ADN par PCR
A, B, C, …, H représentent 8 prélèvements d’ADN testés en PCR. La concentration d’ADN dans ces prélèvements par ordre décroissant est donnée dans le cadre à droite de la figure en unités arbitraires. Le nombre de copies de l’ADN amplifié est fonction du nombre de cycles. On voit qu’après une première phase d’initiation non détectable (bruit de fond), apparaît une phase exponentielle qui se termine par une phase plateau. Le CT (cycle seuil, Cycle Threshold en anglais) est le paramètre déterminé à partir de chaque courbe de fluorescence en fonction du nombre de cycles de PCR. Il représente le nombre de cycles d’amplification à partir duquel la fluorescence est supérieure au bruit de fond, en général d’un facteur 10 (ligne rouge sur le graphe). Plus le niveau d’ADN au départ est élevé et plus le CT est faible. Sur la figure le CT varie de 12 pour le prélèvement A à 35 pour H, indiquant ainsi que la concentration d’ADN du prélèvement A est environ 8 millions de fois (235/212) supérieure à celle du prélèvement H, en supposant que l’efficacité d’amplification est de 2, valeur théorique. (Schéma adapté de Freeman et al., BioTechniques 1999).
C’est à partir des valeurs de CT (nombre de cycles d’amplification à partir duquel l'amplification de l'ADN devient quantifiable; voir figure ci-dessus) qu’on calcule la concentration initiale d’ADN dans le prélèvement de départ (avant amplification). Pour ce faire, on réalise un étalonnage avec une solution d’ADN standard dont on connait la concentration avec précision. Plusieurs dilutions sont réalisées à partir de cette solution et ces diverses solutions sont analysées par PCR. On en déduit une courbe de CT en fonction de la quantité d’ADN standard et on peut évaluer l’efficacité de l’amplification. Celle-ci doit être le plus proche possible de 2 (valeur théorique, puisqu’à chaque cycle de PCR la quantité d’ADN doit doubler). En réalité, l’efficacité de l’amplification notée E est comprise en 1,75 et 2. La courbe d’étalonnage de CT en fonction de la concentration d’ADN standard permet de calculer la concentration d’ADN dans l’échantillon correspondant à la valeur de CT mesurée.
En théorie si N0 représente le nombre de molécules d’ADN dans le prélèvement de départ, on s’attend à ce qu'après n cycles la valeur de N soit égale à N0x2n. Comme indiqué ci-dessus, l’efficacité d’amplification n’est pas de 2 mais d’une valeur E comprise entre 1,75 et 2. On a donc : N=N0xEn.
A partir de là on peut déduire N0 (N0=N/En) le nombre de molécules d’ADN qui étaient présentes dans le prélèvement de départ et donc estimer la concentration de SARS-CoV-2 présente chez le malade.
L’amplification par PCR est énorme et peut atteindre un facteur d’environ mille milliards. La sensibilité très élevée de ce test PCR permet de détecter de très faibles quantités d’ARN viral dans les prélèvements.
Interprétation du test RT-PCR
- Un test RT-PCR positif signifie que le génome (ARN) viral a été détecté dans le prélèvement.
- Ce test RT-PCR évalue la quantité et la seule présence de l’ARN viral dans le prélèvement. Il ne permet pas de savoir si le virus est encore « vivant » ou si le génome détecté est un reliquat inerte du virus. Démontrer que le virus est vivant nécessiterait une inoculation de cellules humaines par un échantillon du prélèvement et la démonstration de la réplication virale au sein de ces cellules. Ce type de test est tout à fait possible en laboratoire de recherche biomédicale (laboratoire de niveau P3), mais ne peut être réalisé en routine.
- La contagiosité et la sévérité de la COVID-19 sont d’autant plus élevées que la charge virale est élevée. La quantité de virus (vivant ou mort) dans le prélèvement est déduite du CT (voir figure ci-dessus). Le CT est généralement compris entre 10 et 40. Plus le CT est faible et plus la charge virale est forte. A l’inverse un CT élevé est le signe d’une charge virale faible. Bien qu’il n’y ait pas aujourd’hui de consensus entre CT et la contagiosité et sévérité de la maladie touchant la personne dont est issu le prélèvement, on estime qu'un CT faible (au-dessous de 25) correspond à une forte contagiosité (charge virale forte), et qu'un CT élevé (supérieur à 30) correspond à une faible contagiosité (charge virale faible). Une valeur du CT supérieure à 40 cycles est considérée comme un résultat négatif (pas d'infection). La valeur du CT n’est malheureusement pas systématiquement donnée par les laboratoires de ville.
- Chez un malade, le test RT-PCR est positif deux à trois jours avant l’apparition des symptômes et jusqu’à 7 à 10 jours après.
- Une personne considérée comme guérie peut rester positive (porteuse de l’ARN viral) pendant plusieurs semaines.
- Chez un porteur asymptomatique le test RT-PCR est positif.
- Un test RT-PCR positif ne signifie donc pas que la personne dont est issu le prélèvement est malade. Il signifie simplement que du génome viral (vivant ou inerte) a été identifié dans le prélèvement.
- Lorsque Santé Publique France annonce par exemple que sur 100 000 tests RT-PCR réalisés, le taux de positivité est de 20%, cela ne signifie pas que 20 000 personnes sont malades. Ce nombre inclut les porteurs asymptomatiques (entre 20 et 50%) et des personnes guéries mais chez qui des reliquats inertes du virus sont encore détectables et non contagieux. Il est donc important de réaliser que les cas positifs en RT-PCR ne reflètent directement pas le nombre des malades.
- Dans tous les cas où le test RT-PCR est positif (malades, porteurs asymptomatiques), un isolement d’au moins une semaine est recommandé pour limiter la transmission.
- Faux positifs et faux négatifs. On estime qu’ils sont très faibles (moins de 1%). Les sources possibles sont les suivantes : erreur dans les tubes de prélèvement, contamination, stockage inadéquat des prélèvement et trop faible quantité de virus dans le prélèvement.
Les tests RT-LAMP sur prélèvements salivaires
Les personnes qui doivent être testées (malades ou pas) redoutent les prélèvements nasopharyngés parfois douloureux. De plus comme je l'ai dit les résultats des tests RT-PCR peuvent prendre plusieurs jours dans des conditions de surcharge médicale car leur réalisation est chronophage et nécessite un équipement sophistiqué. Des tests qui seraient rapides et susceptibles d'être réalisés sans l'utilisation d'une instrumentation trop spécialisée sont donc recherchés. C'est dans ce contexte que les tests salivaires rapides par RT-LAMP (amplification isothermique sans extraction d’ARN) sont en cours d'étude. La mise au point de ce type de test faciliterait les prélèvements (moins désagréable pour le malade), tout en réduisant les risques de contamination pour le personnel préleveur. Par ailleurs, au plan technique, ce type de test basé sur la méthodologie de LAMP serait plus facile à réaliser, avec des temps d'analyse et de lecture des résultats très rapides.
Des publications récentes ont montré que la salive est un outil fiable pour détecter le CoV-2 du SRAS (Azzi et al., J Infect. 2020) avec une forte charge virale chez les patients contagieux. En outre, certaines études ont suggéré que les titres du SARS-CoV-2 dans la salive sont plus élevés que dans les prélèvements nasopharyngés (Wyllie et al., MedRxiv 2020). Des études animales ont montré que le SARS-CoV-2 développe un tropisme cellulaire vers les glandes salivaires, et ceci pourrait donc expliquer cette situation (Chan et al., Clin Infect Dis 2020). Des études comparatives chez l'homme montrent que la présence du SARS-CoV-2 dans la salive peut apparaître plus tôt que dans les prélèvements nasopharyngés. Si on ajoute à cela le fait que la procédure de prélèvement de salive est plus simple pour le soignant et moins invasif pour le patient, le test salivaire présente donc un grand intérêt (To et al., Lancet Infect Dis 2020).
Au plan technique, la méthodologie d'amplification isotherme RT-LAMP est très intéressante car elle permet une transcription inverse rapide de l'ARN (comme dans les tests RT-PCR) mais une amplification de l'ADN sans nécessiter un grand nombre de cycles de montée et descente en température. Un simple chauffage à 65°C pendant moins d'une heure permet de détecter des fragments d'ARN spécifiques. Plusieurs équipes ont déjà utilisé cette méthodologie dans la détection spécifique du SARS-CoV-2 (Tsai et al., Forensic Sci Med Pathol 2020, Ben-Assa et al., Exp Biol Med 2020, Jiang et al., Front Cell Infect Microbiol 2020 ; Yan et al., Clinical Microbiology Infection 2020 ; Lamb et al., Plos One 2020).
Un consortium Montpelliérain (composé de Alcediag et SkillCell deux filiales du groupe Alcen, Sys2Diag un laboratoire CNRS-Alcen, de l'Inserm et du CHU de Montpellier) a développé un test salivaire direct évitant l'extraction d'ARN (L’Helgouach et al. MedRxiv 2020). Il s'agit du test EasyCoV® (https://www.skillcell-alcen.com/products-licensing/easycov-covid-19-salivary-rapid-and-easy-molecular-test).
28 Novembre : Décision de la Haute Autorité de Santé (que je cite ici in extenso):
"Un autre type de tests RT-LAMP sur prélèvement salivaire a été développé, nommé EasyCov®. Il s’agit d’un système « intégré », c'est à dire ne necessitant pas d'intervention de l'opérateur. La sensibilité du test EasyCov® est satisfaisante pour les patients symptomatiques (84 %). En revanche, la spécificité, elle, ne l’est pas : avec 92 %, il est en dessous des performances minimales requises par la HAS (La HAS exige en effet une spécificité de 99% pour les différents types de tests de détection du SARS-CoV2 afin de limiter les faux positifs à moins de 1% des tests réalisés).
Par conséquent, la HAS est favorable à son utilisation et à son remboursement chez les patients symptomatiques pour lesquels le prélèvement nasopharyngé est impossible ou difficilement réalisable. En effet, même si ce test est moins performant que le test RT-PCR nasopharyngé - qui reste le test de référence - cette baisse de sensibilité et de spécificité est compensée par l’acceptabilité et la rapidité de réalisation, avec un résultat en 40 minutes contre plusieurs heures au minimum pour la RT-PCR. Cependant, la moins bonne spécificité impose de réaliser un contrôle par un test RT-PCR sur prélèvement salivaire en cas de test positif."
Enfin, la HAS avait rendu le 18 septembre dernier un avis favorable à l’utilisation des tests RT-PCR sur prélèvement salivaire, dans les cas où les prélèvements nasopharyngés – très invasifs – n'étaient pas possibles ou difficiles.
Les tests antigéniques
Les tests antigéniques ont pour but de détecter la présence du virus dans le prélèvement en mettant en évidence - et éventuellement en quantifiant – une protéine virale (un antigène viral). Ces tests reposent sur la technologie ELISA (Enzyme-Linked ImmunoSorbent Assay) utilisée en routine depuis de nombreuses années dans les laboratoires de recherche biomédicale en immunologie, biologie moléculaire et cellulaire.
Description des tests antigéniques par la technique ELISA
Dans le cadre de la pandémie de COVID-19, c'est généralement la protéine virale appelée nucléocapside (notée N) qui est ciblée car cette protéine est celle qui est la plus fortement représentée dans ce virus et de fait, elle est libérée en grande quantité dans le sérum, les prélèvements nasopharyngés, les échantillons de lavage de gorge, selles et l'urine pendant les premiers jours de l'infection. Sa présence dans le prélèvement permet de poser un diagnostic d'infection par le SARS-CoV-2 en phase précoce. D'autres tests antigéniques ont été décrits et sont réalisés suivant des modalités expérimentales différentes et peuvent cibler des protéines virales différentes (par exemple la protéine Spyke ou l'ARN polymérase ARN-dépendante, NSP12). Cependant le principe de ces tests est similaire à celui décrit ci-dessous.
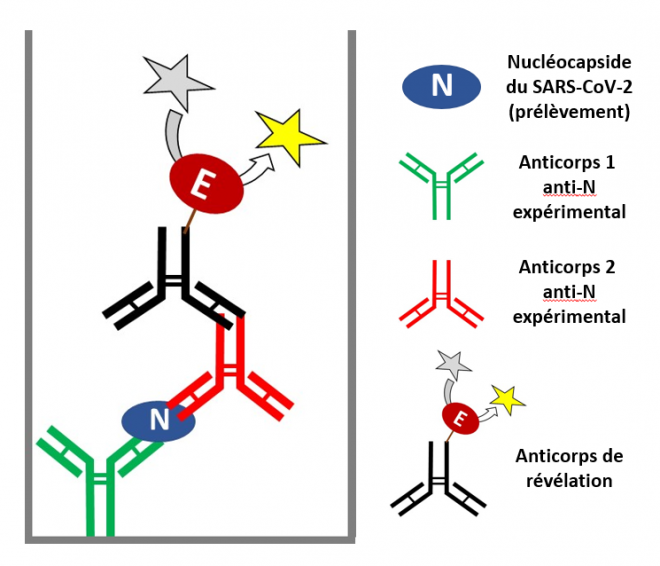
Schéma de principe d'un test antigénique en ELISA
Dans cette procédure un anticorps spécifique contre la protéine virale N (nucléocapside du SARS-CoV-2) a été développé expérimentalement dans un modèle animal (souris, lapin, chèvre, etc.). Cet anticorps (de couleur verte sur le schéma et appelé anticorps 1) est fixé expérimentalement sur le fond du puits réactionnel. Un petit volume du prélèvement (nasopharyngé ou autre) réalisé sur le patient que l'on veut tester est ajouté dans le puits réactionnel. Si le patient est infecté par le SARS-CoV-2, les protéines virales sont présentes dans le prélèvement et notamment la nucléocapside N (ellipse bleue). Cette protéine se fixe sur les anticorps 1 (de couleur verte). On ajoute ensuite à la solution présente dans le puits un deuxième anticorps 2 (de couleur rouge sur le schéma) développé expérimentalement dans un modèle animal (souris, lapin, chèvre, etc.) contre la protéine virale N. Cet anticorps 2 se fixe à son tour sur la protéine N à un épitope différent par rapport à l'anticorps 1. La dernière étape consiste alors à ajouter à la solution un anticorps de révélation. Cet anticorps de révélation (de couleur noire) a été produit expérimentalement dans une espèce animale (rat, lapin, chèvre, etc.) et reconnaît spécifiquement les anticorps (de couleur rouge) dirigés contre la protéine N. De plus, cet anticorps de révélation a été marqué expérimentalement avec un système de signalisation (fluorescence, colorisation, autres). Ici ce système est représenté par une enzyme E (ellipse de couleur bordeaux). Dans la réaction présentée, un substrat non fluorescent ou non coloré (étoile grise) est ajouté au milieu réactionnel et l'enzyme E le transforme en un produit fluorescent ou coloré (étoile de couleur dorée). Si la réaction colorée (ou de fluorescence) se produit, le prélèvement est considéré comme positif. Résultat du test: le patient est infecté. Si dans la même procédure expérimentale l'ajout de substrat (étoile grise) au milieu réactionnel ne produit aucun signal, le prélèvement est considéré comme négatif. Il indique que la protéine virale N n'était pas présente dans le prélèvement. Résultat du test: le patient n'est pas infecté.
Description des tests antigéniques rapides
Les tests antigéniques rapides sont basés sur la méthode ELISA décrite ci-dessus mais utilisent une configuration différente, permettant leur mise en œuvre en pharmacie ou au domicile du patient, sans support technique. De plus leur lecture se fait généralement dans les 15 minutes qui suivent son utilisation.
Le test rapide décrit ci-dessous est basés sur la technique d'immunochromatographie à flux latéral. Il permet de détecter la présence d'une protéine du SARS-CoV-2 (ici la nucléocapside, N) dans le prélèvement réalisé sur une personne suspectée d'avoir contracté le virus. Il se présente sous la forme d'une cassette dont la surface est constituée d'une feuille de nitrocellulose. Ce test utilise la séparation des composants d'un mélange à travers un milieu en utilisant la force capillaire et la liaison spécifique et rapide d'un anticorps à son antigène.
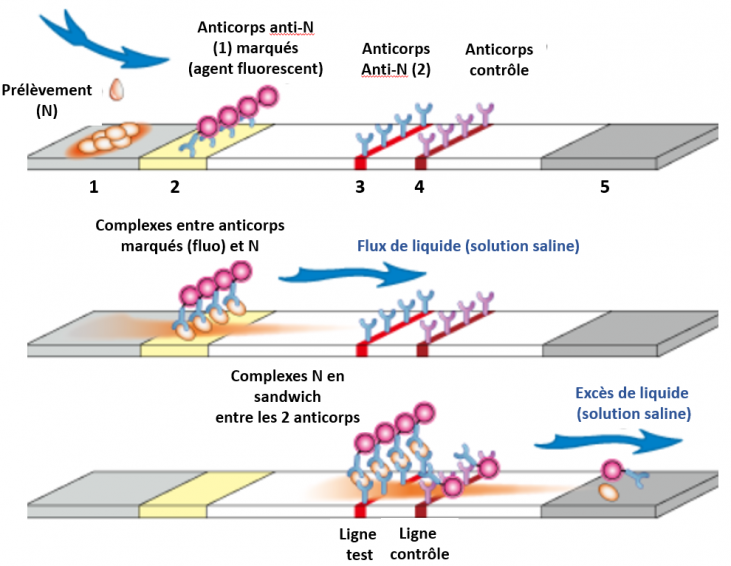
Schéma d'un test antigénique rapide
L'échantillon de prélèvement (nasopharyngé ou autre) contenant la protéine N du SARS-CoV-2 (si la personne est infectée) est appliqué sur la surface 1, dans une solution saline aqueuse. Le liquide migre immédiatement par capillarité vers les autres parties de la feuille de nitrocellulose. Surface 2: un anticorps anti-N (noté 1), développé expérimentalement chez la souris, a été immobilisé. Cet anticorps a été marqué avec un agent fluorescent (ou autre) permettant de l'identifier. Le flux de liquide amène les protéines N au contact de cet anticorps (1) et elles s'y fixent. Les complexes anticorps-protéine N continuent leur migration et arrivent sur la ligne 3. Sur cette ligne est fixé un anticorps anti-N (noté 2) développé expérimentalement chez la souris. Cet anticorps (2) cible sur la protéine N un épitope différent de celui ciblé par l'anticorps (1). Cet anticorps fixe la protéine N des complexes formés sur la surface 2 et il se forme ce que l'on appelle des complexes en sandwich, c'est à dire une protéine N liée à deux anticorps différents. Le flux de liquide continue sa course et les anticorps anti-N (1) résiduels qui n'avaient pas interagi avec la protéine N atteignent la ligne 4 sur laquelle ils se fixent sur des anticorps anti-souris (contrôle). L'intensité de la fluorescence au niveau de la ligne de test correspond à la quantité de la protéine N et est mesurée avec un lecteur de bande optique ou inspectée visuellement. L'apparition de la couleur sur la ligne de contrôle garantit le bon fonctionnement du test. (Schéma adapté de CD Creative Diagnostics).
D’après l’OMS (et selon la Haute Autorité de Santé), les tests antigéniques du SARS-CoV-2 avec une sensibilité ≥ 80 % et une spécificité ≥ 97 % par rapport au test RT-PCR de référence, peuvent être utilisés pour diagnostiquer une infection à SARS-CoV-2 quand les RT-PCR ne sont pas disponibles ou que l’utilité clinique du dépistage serait compromise par des délais trop longs d’obtention des résultats.
Les tests antigéniques ne sont pas recommandés chez les cas asymptomatiques sauf chez les cas-contacts d’un cas confirmé. Par ailleurs, l’OMS ne recommande pas l’utilisation des tests rapides antigéniques pour éviter les faux-positifs.
Ainsi, les tests antigéniques ne sont pas recommandés comme outils de surveillance en conditions de faible circulation du virus. Dans ce cas-là, le test par RT-PCR reste le test de référence.
Les tests sérologiques
Les tests sérologiques sont utilisés pour répondre à la question de savoir si la personne a été infectée par le virus. En effet, si tel est le cas, son système immunitaire a produit des anticorps anti-SARS-CoV-2 (notamment des IgM et IgG). La détection de ces anticorps dans le sang de la personne indique qu'elle a été infectée par le virus, quelle que soit la sévérité de la maladie (grave ou légère), ou même que la personne soit asymptomatique.
Les anticorps IgM et IgG
Avant d'aller plus avant dans la description des tests sérologiques, il faut faire un résumé sur ce que sont les anticorps. Un anticorps (aussi appelé immunoglobuline, Ig) est une grosse protéine en forme de Y utilisée par le système immunitaire pour identifier et neutraliser les agents pathogènes tels que les bactéries et les virus. L'anticorps reconnaît une molécule unique du pathogène, appelée antigène. Un antigène du SARS-CoV-2 est par exemple la protéine de surface spyke (S) ou la nucléocapside N. Chacune des deux extrémités symétriques du Y d'un anticorps contient un site spécifique (aussi appelé paratope) qui va lier une partie spécifique de l'antigène (aussi appelée épitope). Ainsi divers anticorps sont dirigés contre une même protéine sur laquelle ils vont pouvoir se fixer à divers épitopes (c'est à dire diverses parties de la protéine). C'est grâce à ce type de mécanisme de fixation sur les protéines et autres molécules de l'agent pathogène (acides nucléiques, sucres, etc.) que les anticorps vont "signaler" au système immunitaire la présence du pathogène ou d'une cellule infectée par ce pathogène afin qu'il soit neutralisé et détruit par les cellules cytotoxiques (macrophages, neutrophiles, lympocytes T, etc.).
Les deux principales classes d'anticorps sont les IgM et les IgG. Les IgM sont les premiers anticorps sécrétés par le système immunitaire adaptatif en réponse à un antigène étranger. Les IgG sont produits par les lymphocytes B activés. Dans le sang, les IgM se présentent dans une configuration pentamérique (5 molécules d'IgM associées) et ont une demi-vie d'environ 5 à 10 jours. Les principaux anticorps présents dans le sang sont les IgG. Outre le fait qu'ils activent le complément (un groupe de 35 protéines du sang directement impliquées dans les mécanismes d'élimination des pathogènes), ils se lient à des récepteurs spécifiques sur les macrophages et les neutrophiles qui en réponse ingèrent et détruisent les micro-organismes infectieux ainsi "signalés". Les molécules d'IgG sont les seuls anticorps qui peuvent passer de la mère au fœtus via le placenta. De plus, ils sont également sécrétés dans le lait maternel et absorbés par le sang du nouveau-né, protégeant ce dernier contre les infections. En réponse à une infection, les IgM apparaissent après environ une semaine, tandis que les IgG apparaissent plus tardivement, et en général dans les 14 jours, et leur présence peut durer 6 mois, voire plusieurs années, ce qui signifie que les IgG servent d'indicateur d'une infection antérieure. Ainsi, les patients qui sont infectés par le SARS-CoV-2 peuvent être rapidement identifiés par la surveillance simultanée des IgM et des IgG.
Description des tests sérologiques par la technique ELISA
Comme les tests antigéniques, les tests sérologiques reposent sur la technologie ELISA (Enzyme-Linked ImmunoSorbent Assay) utilisée en routine depuis de nombreuses années dans les laboratoires de recherche biomédicale. Voir la figure ci-dessous.
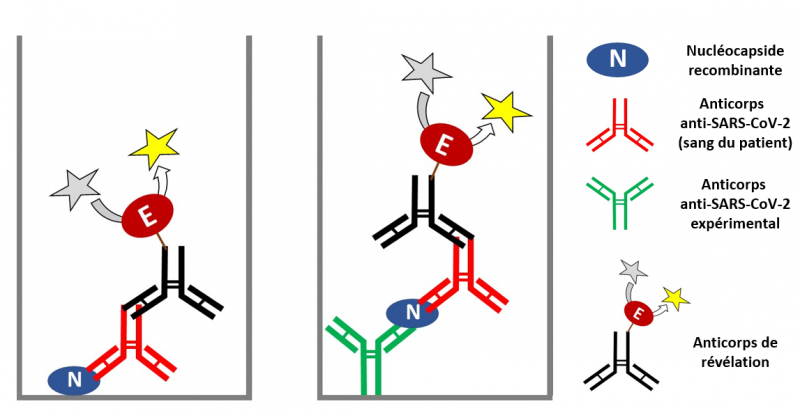
Schéma de principe des tests sérologiques en ELISA
Les tests présentés ici sont réalisés dans un laboratoire de recherche ou un laboratoire d'analyse. Les traits gris figurent le fond et les côtés d'un puits réactionnel (les schémas présentés ne sont pas à l'échelle). Ce puits est rempli d'une solution aqueuse saline (non représentée). Gauche: ELISA simple. Dans cette procédure la nucléocapside N du SARS-CoV-2 (ellipse bleue) contre laquelle on recherche des anticorps dans le sang du patient a été produite par génie génétique (protéine recombinante produite à partir du génome viral) et est fixée expérimentalement sur le fond du puits réactionnel. On ajoute à la solution présente dans le puits un échantillon de sang (ou de sérum) prélevé sur le patients que l'on veut tester. Si le patient a été infecté par le virus, il a développé des anticorps anti-SARS-CoV-2, et ces anticorps (de couleur rouge) vont reconnaître la protéine N et s'y fixer au fond du puits réactionnel. La présence de ces anticorps est révélée par addition d'un anticorps de révélation. L'anticorps de révélation (de couleur noire) a été produit expérimentalement dans une espèce animale (rat, lapin, chèvre, etc.) et reconnaît spécifiquement les anticorps humains (IgG ou IgM). De plus, cet anticorps de révélation a été marqué expérimentalement avec un système de signalisation (fluorescence, colorisation, autres). Ici ce système est représenté par une enzyme E (ellipse de couleur bordeaux). Dans la réaction présentée, un substrat non fluorescent ou non coloré (étoile grise) est ajouté au milieu réactionnel et l'enzyme E le transforme en un produit fluorescent ou coloré (étoile de couleur dorée). Le prélèvement est alors considéré comme positif. Résultat du test: le patient a été infecté. Si dans la même procédure expérimentale l'ajout de substrat (étoile grise) au milieu réactionnel ne produit aucun signal le prélèvement est considéré comme négatif (pas d'anticorps SARS-CoV-2). Résultat du test: le patient n'a pas été infecté. Droite: ELISA "sandwich". Dans cette procédure un anticorps spécifique contre la protéine virale N a été développé expérimentalement dans un modèle animal (souris, lapin, chèvre, etc.). Cet anticorps (de couleur verte sur le schéma) est fixé expérimentalement sur le fond du puits réactionnel. On ajoute à la solution la nucléocapside N du SARS-CoV-2 (ellipse bleue) produite par génie génétique à partir du génome viral, et contre laquelle on recherche des anticorps dans le sang du patient. Cette protéine se fixe sur les anticorps. On ajoute ensuite à la solution présente dans le puits un échantillon de sang (ou de sérum) prélevé sur le patients que l'on veut tester. Si des anticorps anti-SARS-CoV-2 (de couleur rouge sur le schéma) sont présents, ils vont se fixer sur la protéine virale N. La dernière étape consiste alors à ajouter à la solution l'anticorps de révélation (de couleur noire) marqué avec le système de signalisation décrit ci-dessus.
Test de sérologie rapide
Les tests rapides, réalisés en pharmacie ou à domicile sont basés sur la technique ELISA mais réalisés dans une configuration différente (voir figure ci-dessous).
Le test rapide décrit ci-dessous est basés sur la technique d'immunochromatographie à flux latéral. Il permet de détecter la présence d'anticorps anti-SARS-CoV-2 dans le sang (ou le sérum) d'une personne suspectée d'avoir contracté le virus. Il se présente sous la forme d'une cassette dont la surface est constituée d'une feuille de nitrocellulose. Ce test utilise la séparation des composants d'un mélange à travers un milieu en utilisant la force capillaire et la liaison spécifique et rapide d'un anticorps à son antigène. Les IgM et IgG sont des immunoglobulines produites par le système immunitaire pour assurer une protection contre le SRAS-CoV-2 (ou tout autre infection). Les IgM et IgG anti-SARS-CoV-2 peuvent donc être détectées dans les échantillons des patients affectés. L'antigène utilisé dans ce test est la nucléocapside (N) du SARS-CoV-2.
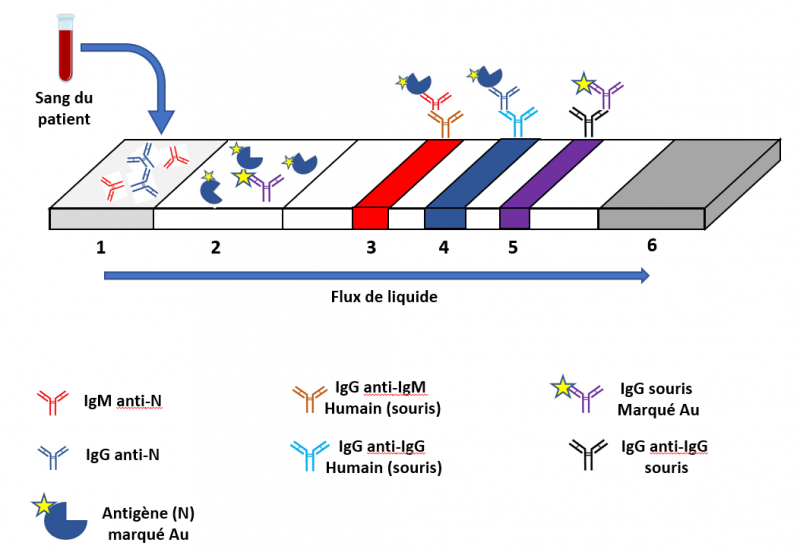
Schéma d'un test sérologique rapide
Surface 1: puits de dépôt du prélèvement (sang ou sérum). Surface 2: réactifs marqués à l'or (Au) colloïdal; dans cet exemple il s'agit de la protéine virale N (nucléocaspisde) marquée à l'or (Au) colloïdal (étoile dorée); cette surface comporte également des IgG de souris marqués à l'or colloïdal; ces anticorps sont utilisés comme contrôle de bon fonctionnement du test. Surface 3: ligne IgM; des anticorps anti-IgM humains ont été fixés sur cette surface. Surface 4: ligne IgG; des anticorps anti-IgG humains ont été fixés sur cette surface. Surface 5: ligne contrôle; des anticorps anti-IgG de souris ont été fixés sur cette surface. Surface 6: absorption du liquide.
Le sang du patient est déposé sur la surface 1. Un petit volume d'une solution aqueuse saline est alors ajouté sur cette surface est se déplace par capillarité vers la surface 6, entraînant avec elle les anticorps et réactifs du test. Si le patient a été infecté, la solution peut contenir des anticorps anti-SARS-CoV-2 (des IgM et/ou des IgG). Dans ce cas, ces anticorps sont entraînés par le flux de liquide comme indiqué sur la figure et ils vont rencontrer l'antigène viral en traversant la surface 2, en l'occurrence la protéine nucléocapside du virus (N marquée à l'or-Au). Les complexe anticorps-antigène se forment et continuent à migrer dans le flux. Ces complexes atteignent la ligne 3. Si des IgM sont présents, ils sont stoppés par les anticorps anti-IgM (comme montré sur le schéma: c'est à dire que ces anticorps fixent les IgM qui ont eux-mêmes fixés la N). Un signal est alors émis sur la ligne 3 révélant la présence d'IgM anti-N dans le sang du patient. Les complexes anticorps-antigène continuent à migrer dans le flux. Ces complexes atteignent la ligne 4. Si des IgG sont présents, ils sont stoppés par les anticorps anti-IgG (comme montré sur le schéma: c'est à dire que ces anticorps fixent les IgG qui ont eux-mêmes fixés la N). Un signal est alors émis sur la ligne 4 révélant la présence d'IgG anti-N dans le sang du patient. Même chose pour les IgG de souris marqués à l'or qui atteignent le ligne 5 où ils sont stoppés par les anticorps anti-IgG de souris. Un signal est alors émis sur la ligne 5 indiquant que le test a fonctionné normalement. La lecture du test est représentée dans la figure ci-dessous.
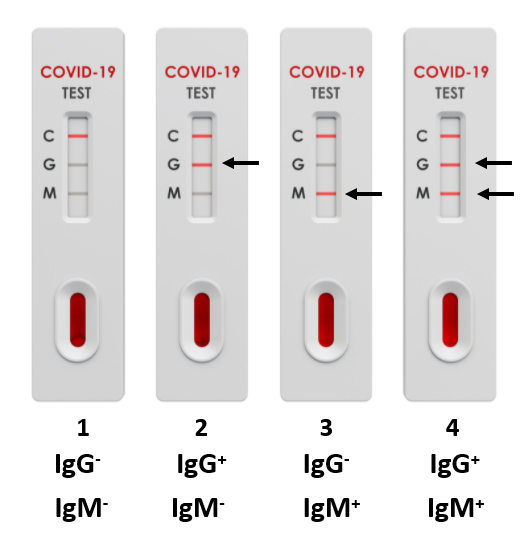
Lecture d'un test de sérologie rapide
1, patient non infecté; 2 infection au SARS-CoV-2 peut-être ancienne (car IgG seuls); 3 infection au SARS-CoV-2 récente (présence d'IgM seuls); 4 infection au SARS-CoV-2 récente (présence des IgM et des IgG). (Schéma adapté de CliniSciences).
Jusqu’à l’avis de la HAS du 2 mai 2020 sur la stratégie d’utilisation des tests sérologiques, la seule technique de diagnostic biologique du COVID-19 recommandée est le test moléculaire par RT-PCR. Cet acte est remboursé y compris lorsqu’il est réalisé en médecine de ville depuis le 7 mars 2020 en fonction de critères établis par la HAS et dans les indications définies et actualisées par les autorités sanitaires.
La surveillance de la séropositivité des anticorps dans une population peut permettre de tirer des conclusions sur l'étendue de l'infection et sur l'incidence cumulative de l'infection dans la population. L'utilisation de la sérologie dans la recherche en épidémiologie et en santé publique permet de comprendre : i) l'occurrence de l'infection dans différentes populations ; ii) combien de personnes sont atteintes d'une infection bénigne ou asymptomatique et qui n'ont peut-être pas été identifiées par la surveillance de routine de la maladie ; iii) la proportion d'infections mortelles parmi les personnes infectées ; iv) la proportion de la population qui peut être protégée contre l'infection à l'avenir.
A suivre...
Date de dernière mise à jour : 15/11/2021
Ajouter un commentaire
