Entrée du virus dans les cellules
Récepteur du SARS-CoV-2 et conséquences thérapeutiques
Résumé
La mise au point de thérapies spécifiques et d’un vaccin contre le SARS-CoV-2, le coronavirus de Wuhan responsable de l’épidémie Covid-19, représente une priorité. Dans cette optique, connaître le mécanisme par lequel le virus entre dans les cellules est fondamental. Les données déjà obtenues sur le SARS-CoV-1 impliqué dans l’épidémie de SARS qui toucha la chine en 2002-2003 et celles obtenues plus récemment sur le SARS-CoV-2, montrent que l’entrée du virus dans les cellules se fait en deux étapes :
Etape 1: Liaison de la protéine virale "spike" (protéine S) à une protéine des membranes cellulaires appelée ACE2 (enzyme de conversion de l’angiotensine).
Etape 2: Activation de la protéine S par des protéases cellulaires dont notamment la protéine TMPRSS2. Cette protéase joue un rôle majeur en activant l’entrée du virus dans les cellules.
Conséquences:
- L'inhibition de TMRPSS2 pourrait représenter une stratégie thérapeutique pour bloquer l’infection Covid-19.
- Des études récentes ont montré qu’un médicament, le Camostat Mesylate, utilisé au Japon pour le traitement des pancréatites chroniques, bloque la protéase TMPRSS2 et pourrait être utilisé pour traiter les malades atteints de Covid-19.
- Un essai clinique va être lancé au Japon pour tester cette hypothèse en Avril 2020. Le Nafamostat Mesylate analogue du Camostat sera également testé.
- La chloroquine exerce son effet antiviral en bloquant l'entrée du virus par la voie des endosomes.
Explications
Nos connaissances actuelles sur le processus d’entrée du SARS-CoV-2 sont largement issues de ce que l’on sait sur le SARS-CoV-1 responsable de l’épidémie de SRAS qui toucha la Chine en 2002-2003 et « cousin » du SARS-CoV-2 (Masters 2006 ; Simmons et al. 2013). Les études toutes récentes menées sur le SARS-CoV-2 (Hoffmann et al. 2020 ; Liu et al. 2020 ; Letko et al. 2020 ; Zhang et al. 2020 ; Yan et al. 2020) confirment et viennent en support de celles réalisées précédemment sur le SARS-CoV-1.
Brève description du virus SARS-CoV-2
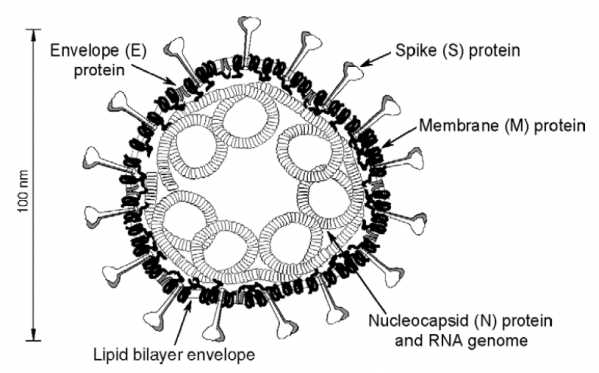
La particule virale a une forme quasi sphérique et un diamètre compris entre 80 et 120 nm (rappel : 1 nm représente un millième de micron qui représente lui-même un millième de mm). Elle est constituée d’une enveloppe elle-même constituée d’une bicouche lipidique (issue de la cellule qui a produit le virus) et de 3 protéines, les protéines S (skipe), M (membrane) et E (enveloppe). Au sein de la particule virale se trouve le génome viral, ARN simple brin positif (c’est à dire codant) étroitement lié à une protéine la nucléocapside (N). L’assemblage nucléocapside-ARN se fait un peu à la façon dont des perles (N) sont attachées sur un fil (l’ARN).
Schéma du virus SARS-CoV (Masters 2006)
Le génome du SARS-CoV-2 (entre 27 et 32 kb) code pour au moins 27 protéines, incluant 15 protéines non-structurales, 4 protéines structurales et 8 protéines dites auxiliaires.
Le virus ne peut se multiplier spontanément à l’extérieur des cellules car il ne possède pas la machinerie nécessaire (ribosomes, ARNs de transfert et nombreux autres agents) pour la synthèse des protéines. Celle-ci lui est fournie par les cellules. L’entrée du virus dans les cellules est donc une première étape essentielle à sa survie.
Entrée du virus SARS-CoV-2 dans la cellule
L’entrée du virus dans la cellule est un processus complexe impliquant deux étapes principales.
Première étape: fixation du virus sur le récepteur
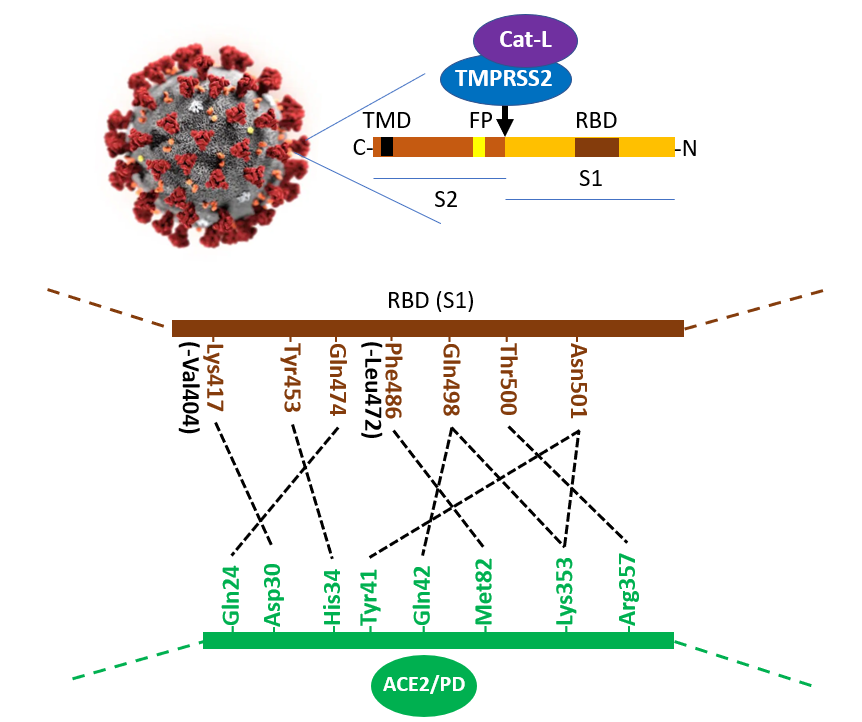
Au cours de la première étape, le domaine de liaison au récepteur (RBD, receptor binding domain) de la protéine S (skipe) se fixe avec une très forte affinité sur le domaine peptidase (PD, peptidase domain) de l’enzyme de conversion de l’angiotensine 2 (ACE2, angiotensin converting enzyme 2), une des protéines de la membrane cellulaire. La protéine S est schématisée dans la figure ci-dessous.
La protéine skype (S) et interaction entre le domaine de liaison du récepteur (RBD) de S et le domaine peptidase (PD) de ACE2.
Partie haute: le virus et vue schématique de la protéine S, montrant le domaine transmembranaire (TMD), le peptide de fusion (FP) et le domaine de liaison du récepteur (RBD). La flèche noire représente le site de coupure de S par les protéases TMPRSS2 et Cathepsine-L (Cat-L).
Partie basse: vue schématique de l'interaction entre le domaine de liaison du récepteur (RBD) de S de SARS-CoV-2 et le domaine peptidase (PD) de ACE2. Les pointillés vert et marron figurent les parties restantes des deux protéines. Les pointillés noirs figurent les interactions entre le RBD et ACE2. Les acides aminés en noir, sont ceux correspondants à la protéine S du SARS-CoV-1 et différents de ceux présents dans la protéine S du SARS-CoV-2. (Yan et al. 2020).
La protéine S est schématiquement divisée en deux parties S1 et S2. La partie S1 de la protéine comprend le domaine de liaison au récepteur (RBD). La partie S2 est impliquée dans l’entrée proprement dite dans la cellule, par un processus de fusion membranaire qui dépend du peptide de fusion (FP, fusion peptide) ; cette partie comporte également le domaine transmembranaire (TMD, transmembrane domain) qui ancre la protéine S dans la membrane virale.
La protéine ACE2 est présente dans le poumon (pneumocytes et macrophages), le cœur, l’intestin et le rein. La forte affinité de fixation du virus sur ACE2 résulte d’interactions polaires et de ponts hydrogène entre des acides aminés de la protéine virale S et des acides aminés de la protéine cellulaire ACE2. La figure ci-dessus (partie basse) montre les acides aminés impliqués dans cette fixation (Yan et al. 2020).
Lorsqu’on compare la séquence du RBD du virus SARS-CoV-1 avec celle du SARS-CoV-2 des différences apparaissent. Rappelons que l’homologie de séquence pleine longueur en acides aminés entre les deux coronavirus est de 94,6%. Notamment dans le SARS-CoV-1 les acides aminés Val404 et Leu472 remplacent les Lys417 et Phe498 du SARS-CoV-2 (Yan et al. 2020). On comprend ainsi pourquoi l’affinité de liaison entre les deux virus et la protéine ACE2 sera différente. Or, l’affinité entre la protéine virale et la protéine récepteur humaine (ACE2) joue un rôle déterminant dans la transmissibilité du virus. Si pour un coronavirus d’une espèce animale X cette affinité avec ACE2 est faible (du fait de différences dans la séquence du RBD), il y a peu de chance que l’espèce X soit responsable de la transmission à l’homme. C’est le cas du coronavirus MERS (transmis par le chameau) qui utilise en fait un autre récepteur, différent de ACE2. C’est sur cette base que se pose la question de savoir quelle est l’espèce animale relai entre la chauve-souris et l’homme dans l’infection. C’est également sur cette base que le pangolin avait été suspecté être cette espèce animale. En effet, l’homologie de séquence du domaine de liaison (RBD) au récepteur du coronavirus du pangolin est 99% identique à celle du SARS-CoV-2 (Xiao et al. 2020). Cependant, d’autres études ( Liu et al. 2020, Zhang et al. 2020, Lam et al. 2020) ont ensuite montré que si on considère la séquence pleine longueur (et non seulement la séquence du RBD) du coronavirus qui infecte les pangolins de Malaisie, l’homologie de séquence n’est plus que de l’ordre de 85 à 92% et cela suggère donc que la source du SARS-CoV-2 ne peut être cet animal.
Deuxième étape: activation de l'entrée cellulaire
Bien que l’ACE2 soit présente dans les cellules endothéliales de tous les organes, le SARS-CoV est essentiellement pathogène pour le poumon. De plus, le tropisme cellulaire du virus ne corrèle pas strictement avec la distribution de ACE2. Par exemple, alors que l’expression de ACE2 se fait dans les pneumocytes 1 et 2, seuls les pneumocytes 1 impliqués dans les échanges d’oxygène sang-tissu au niveau des alvéoles pulmonaires sont infectés par le virus. Cela suggère que le récepteur ne peut agir seul et qu’un cofacteur est nécessaire pour l’entrée du virus dans les pneumocytes (Kawase et al. 2012).
Pour le SARS-CoV, ce rôle de cofacteur est tenu par au moins deux protéases, la TMPRSS2 (transmembrane serine protéase) et la cathepsine-L (une cystéine protéase).
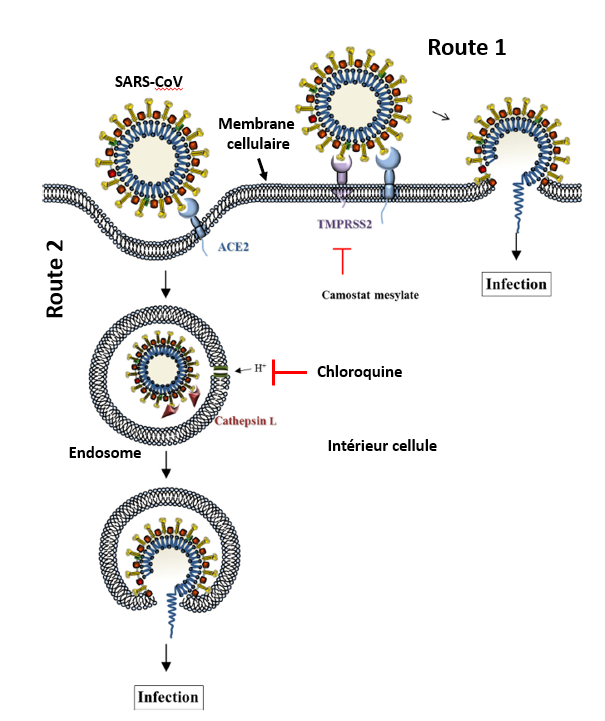
Lorsque la protéine S est fixée sur l’ACE2, un changement de conformation de la protéine se produit, rend celle-ci sensible aux protéases TMPRSS2 et/ou cathepsine L qui par coupure éliminent la partie S1. Il s’agit d’un processus d’activation protéolytique de la protéine virale S qui « découvre » le peptide de fusion FP. C’est ce peptide FP qui, rendu accessible, permet l’entrée proprement dite du virus dans la cellule soit par un processus de fusion entre la membrane virale et la membrane cellulaire après activation par la TMPRSS2, soit par un processus de fusion entre la membrane virale et la membrane des endosomes cellulaires après activation par la cathepsine-L (Simmons 2013). Ce processus se termine par l’introduction du génome viral dans la cellule comme indiqué dans la figure ci-dessous. Il semble que ces deux processus ne soient pas mutuellement exclusifs et agissent simultanément.
A noter que la chloroquine reconnue pour son effet antiviral sur le SARS-CoV-2 (Wang et al. 2020 et Gao et al. 2020) exercerait son action en augmentant le pH dans les endosomes, réduisant ainsi l'activité de la cathepsine-L et inhibant la fusion de la membrane virale et de la membrane des endosomes.
Schéma du mécanisme d'entrée du virus SARS-CoV dans les cellules (selon Simmons 2013)
La libération du génome viral dans la cellule peut se faire suivant deux routes différentes, suivant la protéase utilisée.
Route 1 : activation de la protéine virale S par la protéase TMPRSS2. La protéine TMPRSS2 est exprimée sur les membranes comme la protéine ACE2. Ces deux protéines cohabitent et la liaison de la protéine S du virus sur ACE2 suffit pour que TMRPSS2 coupe S, exposant ainsi le peptide de fusion FP. C’est par l’intermédiaire de FP que la membrane du virus fusionne avec celle de la cellule. La particule virale ainsi ouverte peut libérer son contenu, c’est à dire le génome viral, dans la cellule.
Route 2 : activation de la protéine virale S par la cathepsine-L. Les endosomes se forment par invagination de la membrane cellulaire, suite à la liaison du virus sur son récepteur. Dans les endosomes, le pH diminue par entrée de protons. Ce processus conduit à l’activation de la cathepsine-L qui peut alors couper et activer la protéine S du virus, exposant ainsi le peptide de fusion FP. C’est par l’intermédiaire de FP que la membrane du virus fusionne avec celle de l’endosome. La particule virale ainsi ouverte peut libérer son contenu, c’est à dire le génome viral, dans la cellule.
Il est possible que d’autres protéases soient également impliquées dans l’activation de la protéine virale S (Simmons 2013).
Conséquences thérapeutiques
Les études décrites ci-dessus montrent clairement comment le clivage protéolytique de la protéine virale S est essentiel pour l’entrée du virus dans les cellules. La connaissance de ce mécanisme ouvre la voie à la mise en place de stratégies thérapeutiques. Ces stratégies visent les acteurs essentiels de l’entrée virale, c’est à dire les protéases TMPRSS2 et cathepsine-L. Du fait du rôle central que joue la cathepsine-L dans le fonctionnement des endosomes, son inhibition risque de provoquer des effets délétères et une application clinique semble pour le moment peu probable. Mais l'inhibition indirecte de cette protéase par la chloroquine qui augmente le pH à l'intérieur des endosomes, semble avoir un effet bénéfique sur l'infection Covid-19 (Service D Raoult, CHU La Timone). Pour ce qui concerne la protéase TMPRSS2, il existe déjà un médicament utilisé au Japon pour le traitement de la pancréatite chronique, le camostat mesylate, qui bloque l’activité biologique de la TMPRSS2 (Kawase et al. 2012 ; Zhou et al. 2015; Hoffmann et al. 2020). Ce médicament et certaines molécules apparentées (notamment le nafamostat mesylate) aux mêmes effets thérapeutiques pourraient être utilisés pour le traitement des malades infectés par le SARS-CoV-2 dans les prochaines semaines (https://www.nippon.com/en/news/yjj2020031800741/pancreatitis-drug-may-block-coronavirus-infection-u-of-tokyo.html).
De plus, on sait que les malades guéris de l’infection SARS-CoV-1 possèdent des anticorps neutralisants qui peuvent être détectés deux ans après l’infection et qui sont dirigés contre le protéine virale S. Il a été récemment montré que ces anticorps sont capables de bloquer l’infection in vitro de cellules humaines par le SARS-CoV-2 (Hoffmann et al. 2020). Un vaccin dirigé contre la protéine virale S du SARS-CoV-1 pourrait donc être efficace pour une protection contre le SARS-CoV-2.
Date de dernière mise à jour : 14/11/2020
Commentaires
-

- 1. Rochefort Le 28/05/2022
Merci Patrick, très pédagogique et utile.
Henri -

- 2. Alain chavanieu Le 23/03/2020
Merci Patrick pour ces résumés.
Alain
Ajouter un commentaire